Nat. Mach. Intell. | 探索稀疏化学空间的化学语言模型新策略
今天给大家介绍来自不列颠哥伦比亚大学和阿尔伯塔大学联合发表的一篇文章。该文章系统地评估并优化了基于循环神经网络在低数据环境中的分子生成模型。发现该模型可以从更少的例子中学习到健壮的模型。同时,本文还确定了低数据下,得到等学习效果和等质量模型的策略;特别是通过枚举非规范SMILES进行数据增强,并通过学习细菌、植物和真菌代谢组模型来证明这些策略的可用性。并且,本文还对评估生成模型的指标进行了基准测试,发现该领域中许多最广泛使用的指标未能捕获模型质量,同时确定了一些表现良好的指标。

1
介绍
目前人类已经探索出了大量分子,即便如此,这些分子在广阔的化学空间中也仅仅占一小部分,就目前的医学实践与无限的可能性来说,更有效的化学空间导航(分子发现)方法或能帮助解决人类面临的各种紧迫挑战。
最近,深度生成模型已成为化学空间探索的强大工具。这些模型利用深度神经网络来学习隐含在训练分子中的化学物质。一旦经过训练,这些模型就能够从目标化学空间中随机采样没见过的分子。
许多成功的生成模型方法都是学习生成分子的文本表示,通常采用简化的分子输入行输入系统(SMILES)格式(图 1a)。基于 RNN (图 1b)的 SMILES 字符串模型表现较优,本文将其称为化学语言模型( CLM)。
CLM因为其“逆向设计”(生成需要特性的分子)的可行性引起了人们的兴趣。其中有一个突出的挑战——需要大量的训练数据(数十万到数百万数量的分子),然而通常情况下,目标探索化学空间并没有相应数量的示例。为了在低数据情况下实现生成建模,已经开发了基于强化学习 (RL)或迁移学习 (TL) 的方法,即模型首先在大型通用化学结构数据库上进行“预训练”,然后进行第二轮“微调”,旨在缩小在有更多约束的化学空间。然而它们都有模式崩溃和灾难性遗忘的缺点。理想情况下是可以直接从少量示例中学习生成模型。本文则从这方面着手展开了研究。
2
结果
首先确定训练强大的 CLM 所需的最小分子数。随机在ZINC 数据库中抽取1,000 到 500,000 个 SMILES 字符串样本来训练模型,每个训练模型中采样 500,000 个 SMILES(图 1c)。计算了每个模型生成的有效 SMILES 的比例,随着训练集大小的增加,有效分子的比例迅速增加,在大约 50,000 个分子后迅速饱和(图 1d)。
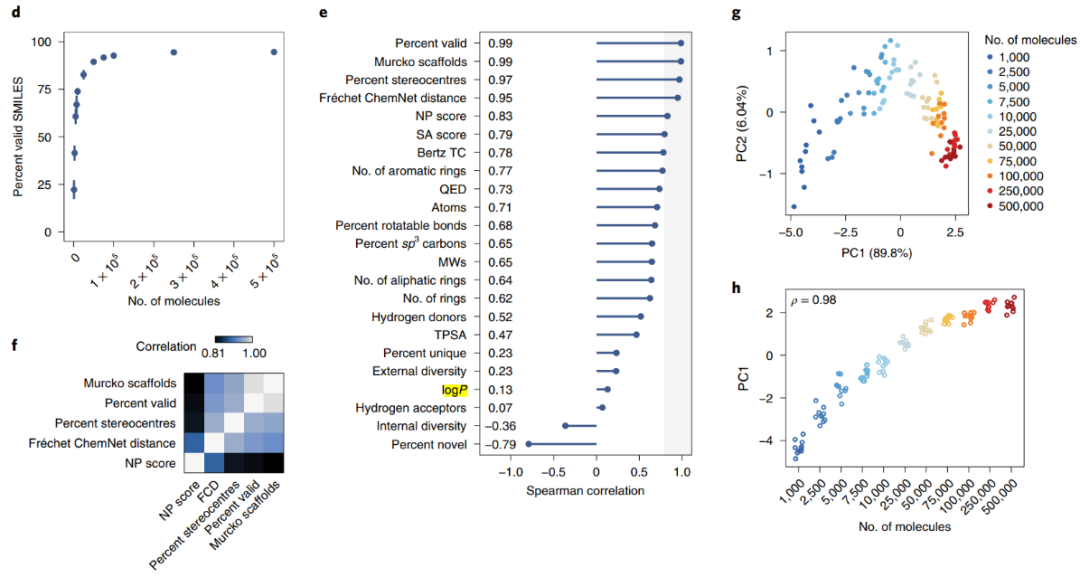
图1 从有限数据中学习生成分子
广泛使用的指标无法捕获生成模型的性能
模型可能已经学会了生成有效的 SMILES 字符串,但生成的分子与训练集中的分子几乎没有相似之处。因此,我们寻求对模型性能进行更全面的评估。
作者选取了23个曾被提议用于评估分子生成模型的指标,并推断随着训练集大小的增加,指标度量得到的模型性能也增加,将该推断作为本实验的“ground truth”。通过计算训练集大小与每个度量值之间的 Spearman 等级相关性来比较此23个指标。令人惊讶的是,23 个指标的相关性表现出巨大差异(图 1e),只有少数指标与训练集的大小密切相关,大多数与这个实验的“ground truth”充其量仅仅是适度相关,其中还有两个最广泛使用的指标:Percent unique和log P。
分子生成模型的整体评估
整合几个表现最佳的指标,以得出模型性能的单一衡量标准。然而,这些指标是在非常不同的尺度上测量的,并表现出复杂的相关性(图 1f)。主成分分析 (PCA) 中,用“ground truth”选择主成分PC1(图 1g,h)。此外,整合多个指标后在数据集超过100万个分子后模型性能继续提高,说明CLM 首先学会产生有效的 SMILES,然后才学会匹配目标分子的结构和物理化学特性。因此,整合多个不同的指标对于整体评估是必要的。
学习不同化学空间的 CLM
从三个其他数据库(具有不同的结构特性)中采样分子(图 2a),重复前面实验,本文还测试了强大的 CLM 所需的分子数量是否会随着目标化学空间的变化而变化。加上前面的ZINC数据库,数据库的分子复杂程度从高到低依次为COCONU、ChEMBL、ZINC 和 GDB(图 2b)。图 2c显示,学习健壮模型所需的最少示例数量取决于目标化学空间的复杂性。23个指标的实验与ZINC数据集大同小异(图 2d);在每个数据库中分别执行 PCA 时获得了类似的结果(图 2d-f)。图 2h表明从 GDB 数据库获得的结果可能不适用于更复杂分子模型。
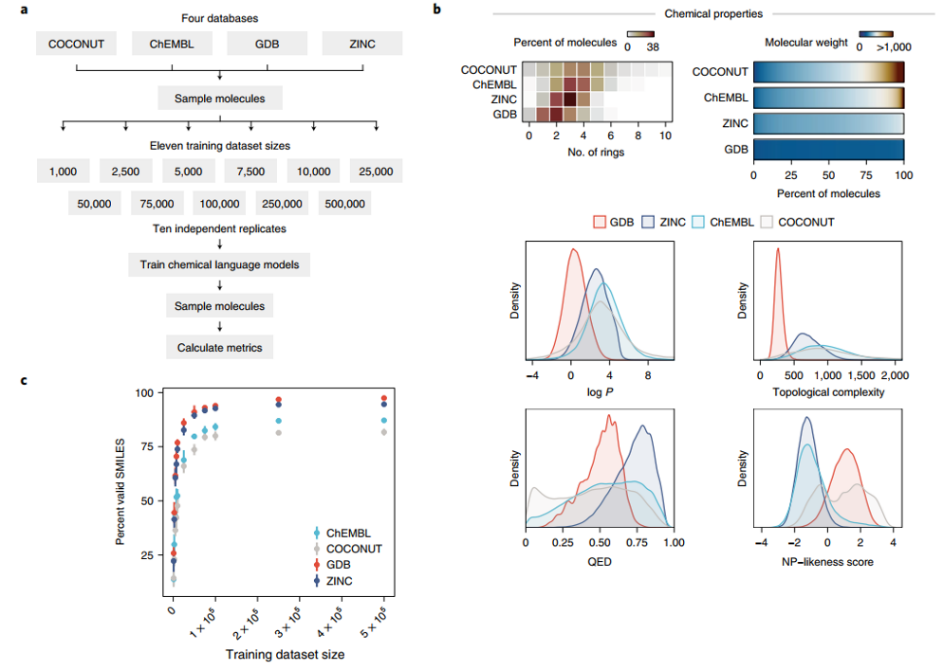
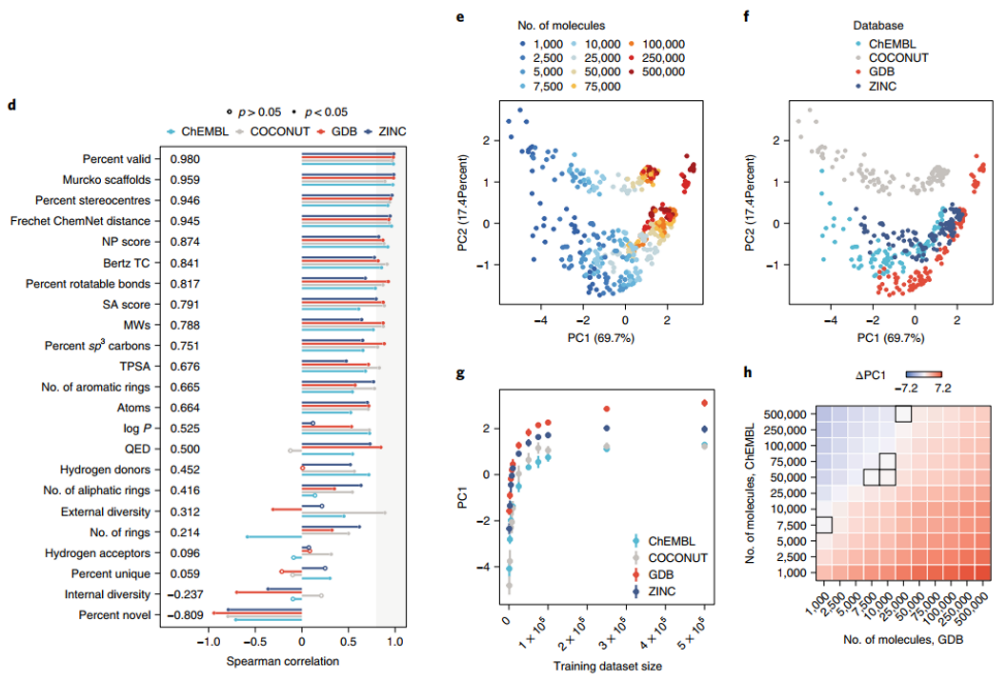
图2 不同化学空间的少量数据生成模型
单独指标和 PC1 分数都表明性能随着采样分子多样性的增加而下降(图 3)。这些发现表明,从少量示例中学习 CLM在化学空间相对同质的区域中更有可能取得成功。
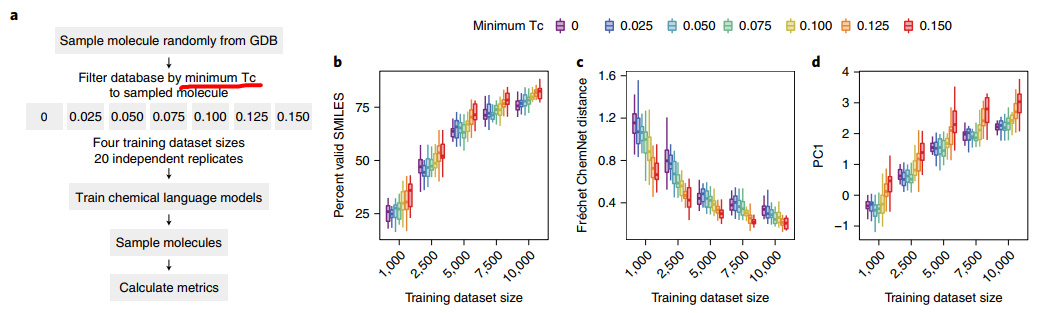
图3 多样化和同质分子的少量数据生成模型
评估 CLM 的分子表征
SMILES 字符串训练的模型通常会产生很大比例的无效分子,DeepSMILES 变体对 SMILES 语法进行了两项修改,以消除与环和分支表示相关的长期依赖关系。自引用嵌入字符串 (SELFIES) 是基于 Chomsky type-2 语法的完全不同的表示,其中每个 SELFIES 字符串指定一个有效的化学图 。
在四个数据库的分子SMILES、DeepSMILES 和 SELFIES 表示上训练了生成模型(图 4a)。在 SELFIES 字符串上训练的模型确实以 100% 的比率生成了有效的化学图(图 4b)。而在 DeepSMILES 上训练的模型并没有比在规范 SMILES 上训练的模型更快速地产生有效分子。
为了研究在每个表示上训练的模型如何学习匹配目标化学空间,再次执行 PCA。然而作者发现训练生成 SELFIES 字符串的模型始终比在相同分子的 SMILES 或 DeepSMILES 表示上训练的模型获得更低的 PC1 分数(图 4c )。检查单个指标也证实了这一趋势:例如,在 SELFIES 上训练的模型与训练集的 Fréchet ChemNet 距离也更高(图 4d)。结果表明,过滤掉无效分子后,在 SMILES 字符串上训练的模型比在替代表示上训练的模型更匹配目标化学空间。
数据增强对 CLM 的矛盾影响
按照惯例,每个化学结构都有一个单一的、“规范的” SMILES 表示。然而,通过改变分子中原子的遍历顺序,也可以列举数百个“非规范”SMILES 表示(图 4h)。非规范 SMILES 的枚举已被用于通过训练序列到序列模型来学习化学结构的连续表示,并且最新研究表明 SMILES 枚举可以提高生成模型的质量。
作者测试了 SMILES 枚举是否可以减少学习 CLM 所需的训练示例数量(图 4h)。在枚举 SMILES 上训练的模型以显着更高的速率生成有效分子,尤其是在最小的训练数据集中(图 4i)。
PCA 强调了 SMILES 枚举的上下文特定影响(图 4j)。数据增强对非常小的训练集学习的模型的效果最好;在很大的训练集中甚至偶有负面影响(图 4k)。这表明,数据增强最适合用于低数据区域,尤其是结构复杂的分子。
量化SMILES 枚举的性能影响发现(图 4l、m),在结构化的大型数据集中存在“过度枚举”的可能性,反映了SMILES 枚举的矛盾影响。
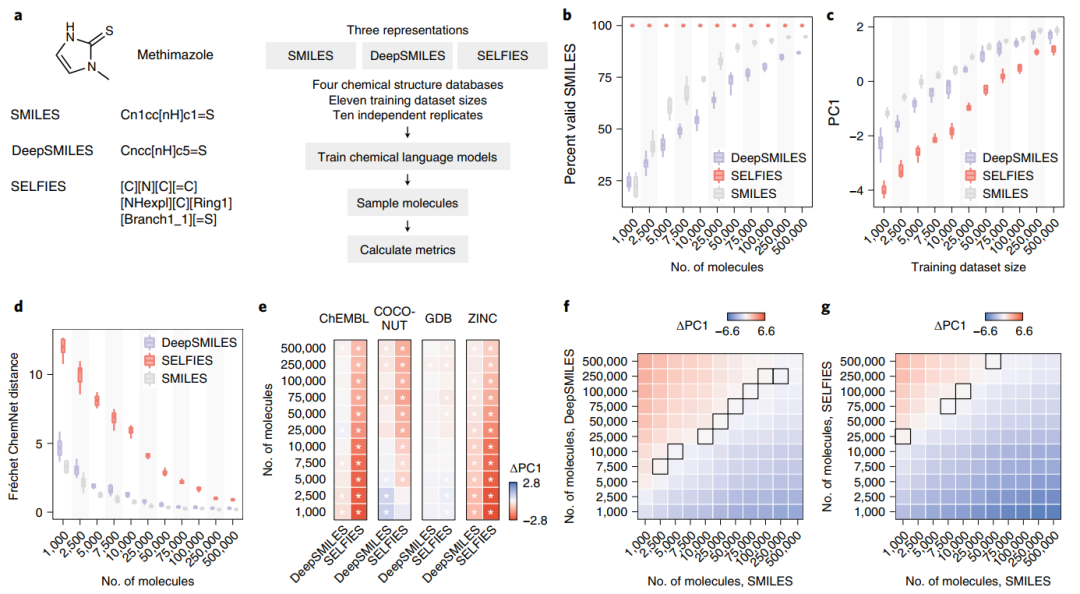
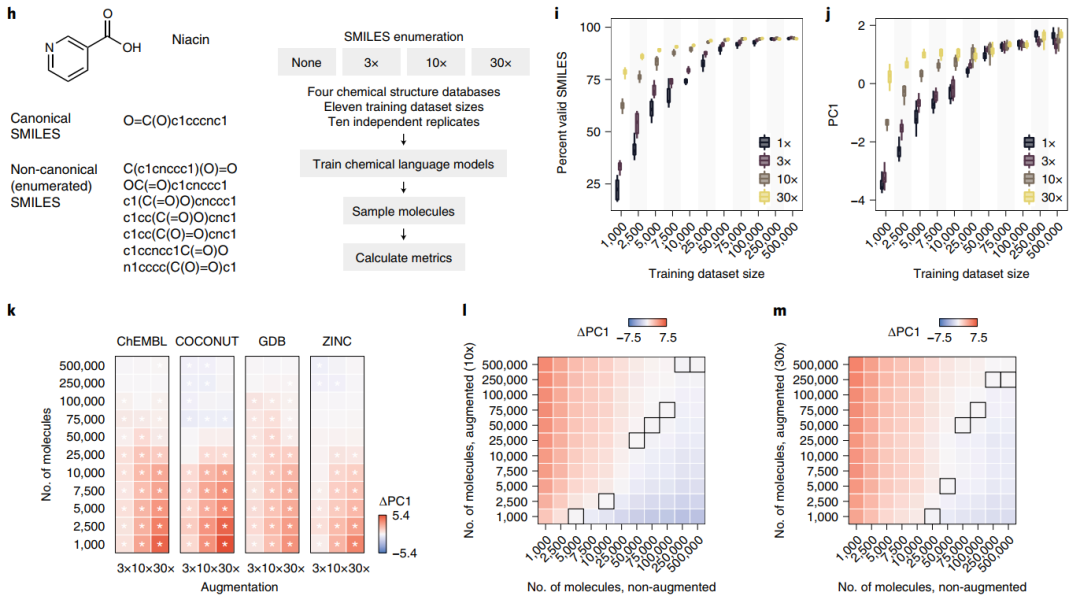
图4 少量数据生成模型的替代分子表示
数据而非架构决定了低数据模式下的模型性能
图 5显示出CLM 训练集的重要性,在大型超参数网格中,超参数的调整几乎不能像增加训练集的大小那样影响性能。
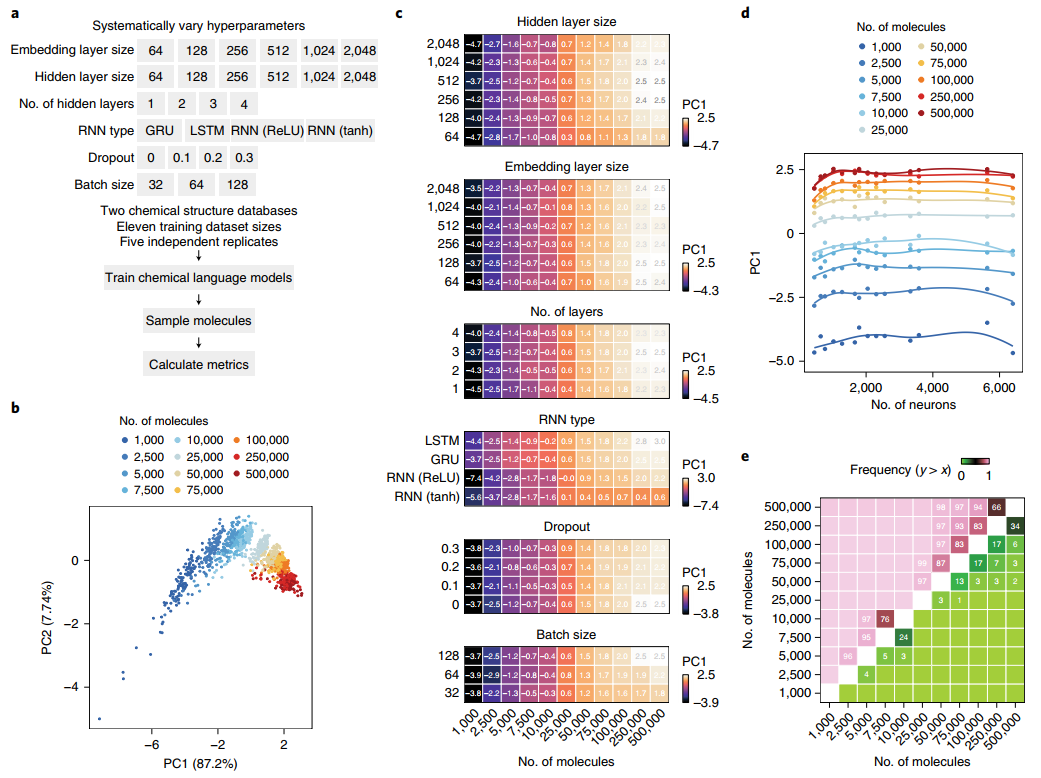
图5 数据决定少量数据生成模型的性能
学习细菌、真菌和植物代谢组的生成模型
上文阐明了从有限的训练数据中学习 CLM 的原则,作为原则的阐述,作者组装了细菌、真菌和植物代谢物的数据库,但每个数据库仅包含 15,000-22,000 个分子(图 6a)。使用具有高度 SMILES 枚举的 LSTM 作为最佳策略(图 6b),尽管训练数据有限,但优化后的模型生成的分子的理化特性与目标代谢组的物理化学特性非常匹配;此外,生成模型几乎完美地再现了三个目标代谢组的化学空间(图 6c)。综上所述, CLM 可以直接从少量训练示例中学习,重现甚至非常复杂的化学空间。
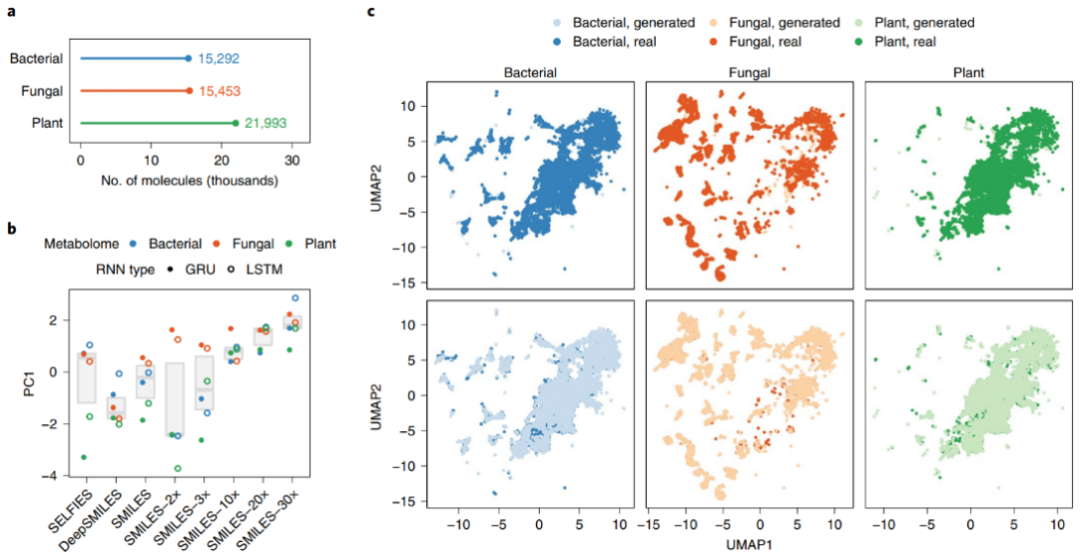
图6 细菌、真菌和植物代谢组的少量数据生成模型
3
方法
输入数据 实验从四个化学结构数据库中学习分子的生成模型:ZINC 数据库;GDB-13 数据库,它列举了所有可能的包含13 个原子的有机小分子;ChEMBL 数据库,包含具有类似药物特性的生物活性小分子;天然产物 COCONUT 数据库。
化学语言模型 除非另有说明,语言模型的架构由三层 GRU 组成,其中隐藏层为 512 维,嵌入层为 128 维,没有 dropout 层。使用 Adam 优化器训练模型,β1 = 0.9 和 β2 = 0.999,批量大小为 128(除非另有说明)和学习率为 0.001。
评估模型性能 Spearman 等级相关性来考虑23个评估指标与数据集大小的关系。结合五个表现最佳的指标的信息,同时考虑指标之间的协方差,使用 R 函数“princomp”执行 PCA。
代谢组的生成模型 为了训练细菌、真菌和植物代谢组的生成模型,作者编译了几个已知代谢物数据库:大肠杆菌代谢组数据库 (ECMDB)、铜绿假单胞菌代谢组数据库 (PAMDB)、StreptomeDB、NPASS和 BioCyc。在前三个代谢组上训练了总共 48 个化学生成模型。
4
总结
CLM 已成为探索化学空间的强大工具。然而,人们普遍认为这些模型需要非常大型的训练集。在本文中,作者量化学习强大的 CLM 所需的最少分子数量,并确定降低此下限的策略。为在稀疏化学空间区域直接学习生成模型提供了基础。
参考资料
Skinnider, M.A., Stacey, R.G., Wishart, D.S. et al. Chemical language models enable navigation in sparsely populated chemical space. Nat Mach Intell (2021).
https://doi.org/10.1038/s42256-021-00368-1
代码
http://github.com/skinnider/low-data-generative-models
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/120559057
- 点赞
- 收藏
- 关注作者


评论(0)