张小白带你体验昇思1.5的MindScience

唉,其实说起昇思,张小白也是一头雾水。叫了1年多的MindSpore“头孢”, 被以“上不得厅堂”为由,强行改为“昇思”。其实Mind是头脑,Spore是孢子。头孢这个词真的信达雅的翻译,而且,专治各种AI的不服,跟头孢作为药物具备广谱杀菌的超高实用价值,其实也是对应的。
不过领导的话还是不能不听的,你说叫啥就叫啥吧。张小白宁可偷偷地叫它“曼sir跑",想必大姐大应该不会有什么想法。
既然自称不喜欢4,跳过了1.4版本直接推出1.5版本,但是请告诉我为啥它自己要叫”昇4“?有深思,还有熟虑吗?
以上是对1.5版本名字的吐槽,张小白对昇思已经熟虑过了,就到此为止了。
------------------------------华丽的分割线----------------------------------
张小白曾经在微信群里面问大家,1.5版本有啥好体验的?AI大侠姐夫甲的四弟姐夫丁建议说:要不试试MindScience?

沿着丁大大指引的方向走路一定是没错的。
既然张小白曾经用了WSL装了GPU版本的MindSpore( https://bbs.huaweicloud.cn/blogs/293332 ),不如就这样一路走下去吧。
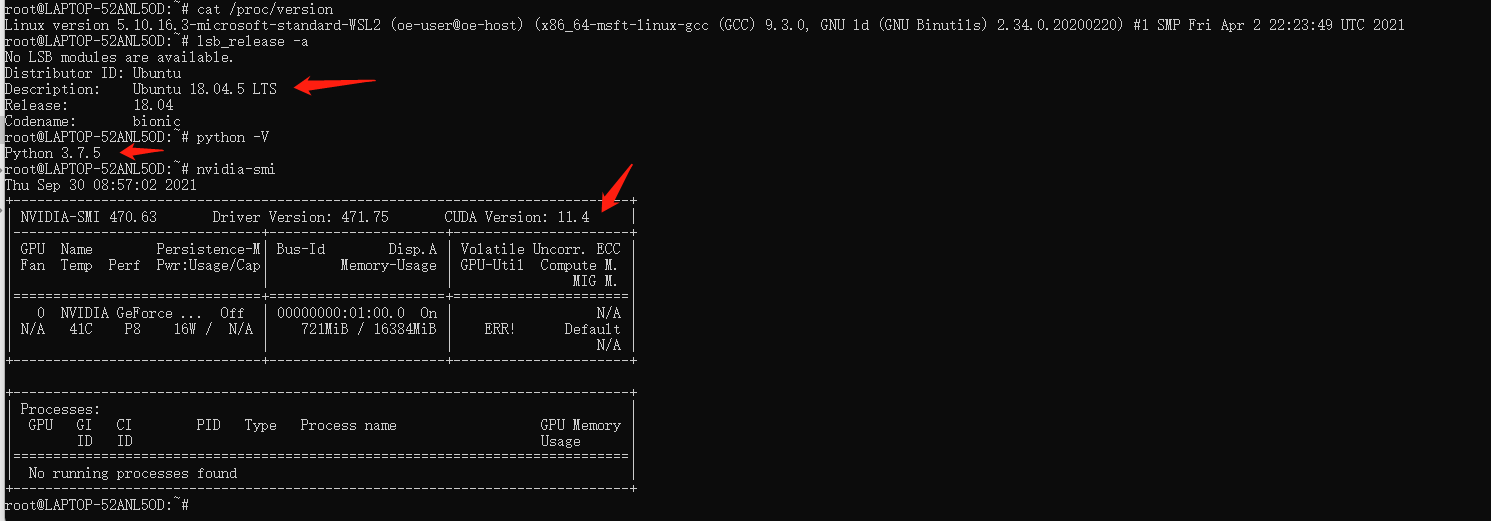
先看一下现在的环境:
Ubuntu 18.04
Python 3.7.5
CUDA 11.4
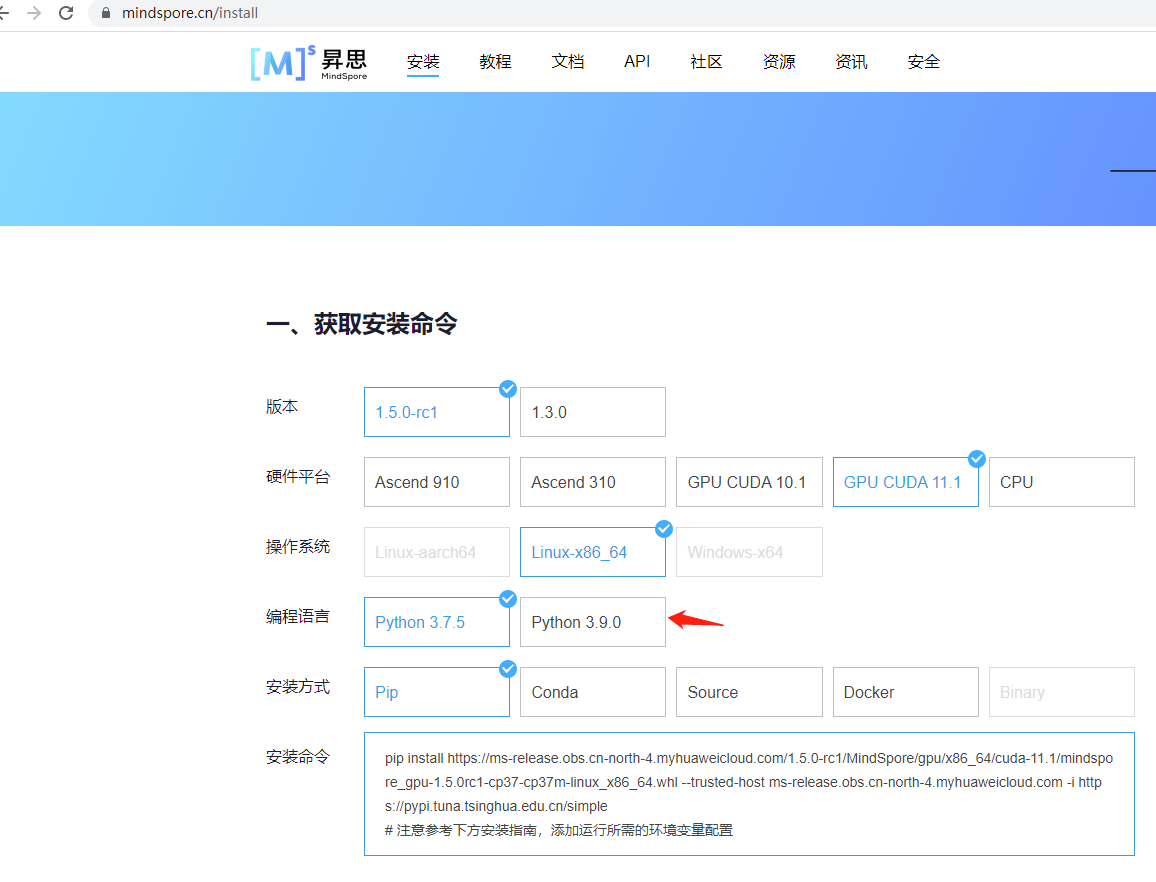
从官方文档上来看,这次MindSpore 1.5的升级不仅可以支持Python 3.7.5,还可以支持Python 3.9.0了。
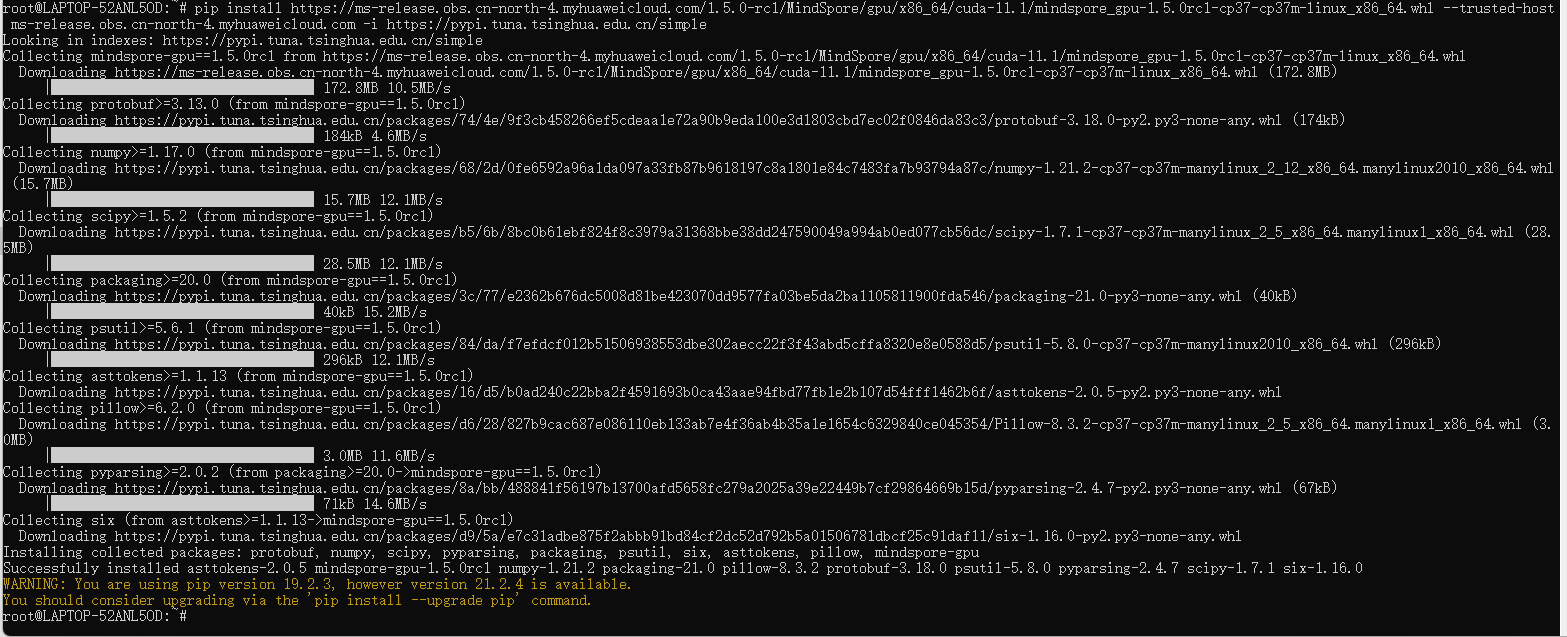
那就直接装起来吧:
pip install https://ms-release.obs.cn-north-4.myhuaweicloud.com/1.5.0-rc1/MindSpore/gpu/x86_64/cuda-11.1/mindspore_gpu-1.5.0rc1-cp37-cp37m-linux_x86_64.whl --trusted-host ms-release.obs.cn-north-4.myhuaweicloud.com -i https://pypi.tuna.tsinghua.edu.cn/simple
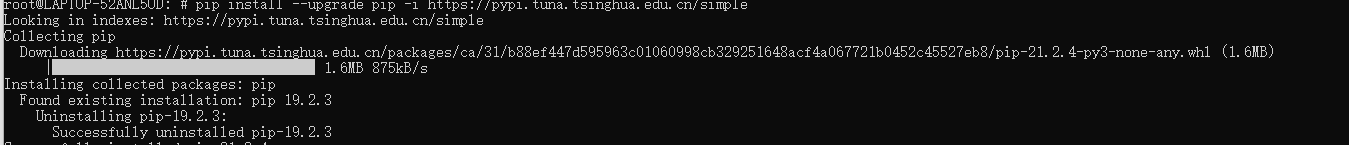
在做别的事情之前,先根据提示升级下pip:
pip install --upgrade pip -i https://pypi.tuna.tsinghua.edu.cn/simple
根据官方文档,试验第一种验证方式:
python -c "import mindspore;mindspore.run_check()"
再试验第二种验证方式:
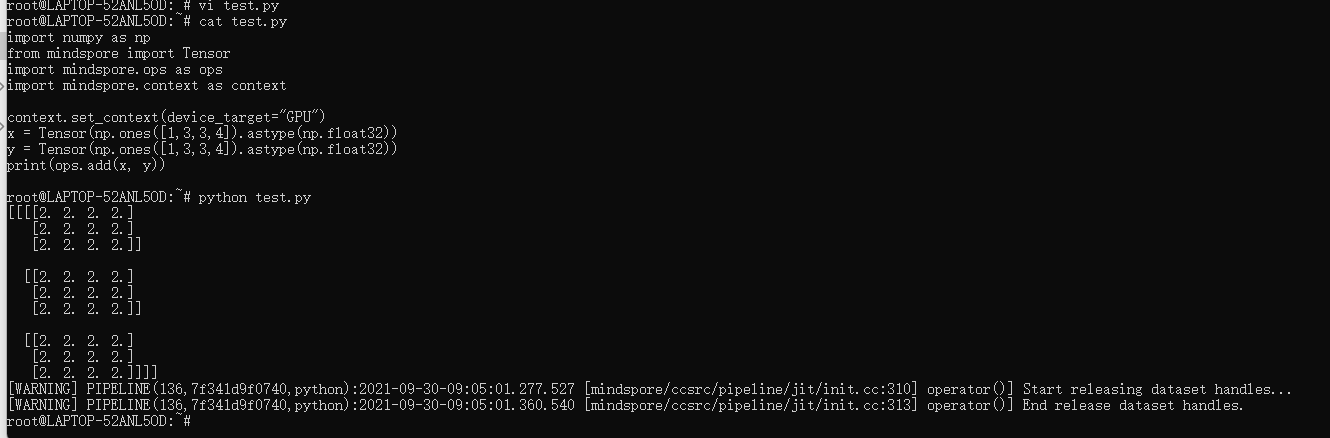
看来没啥问题。GPU表现良好,不愧是张小白的3080移动版显卡。(具体显卡折腾过程参见:https://bbs.huaweicloud.cn/forum/thread-146553-1-1.html )
这次MindSpore 1.5特意提到,它不想仅仅做一个AI的框架了,它还想具备科学计算的能力。(这充分说明,能做AI的它已经很AI了。。。)所以,物理、化学、电子信息、生物、材料。。。。这些学科中如果需要科学计算的,没准都能用上MindSpore:
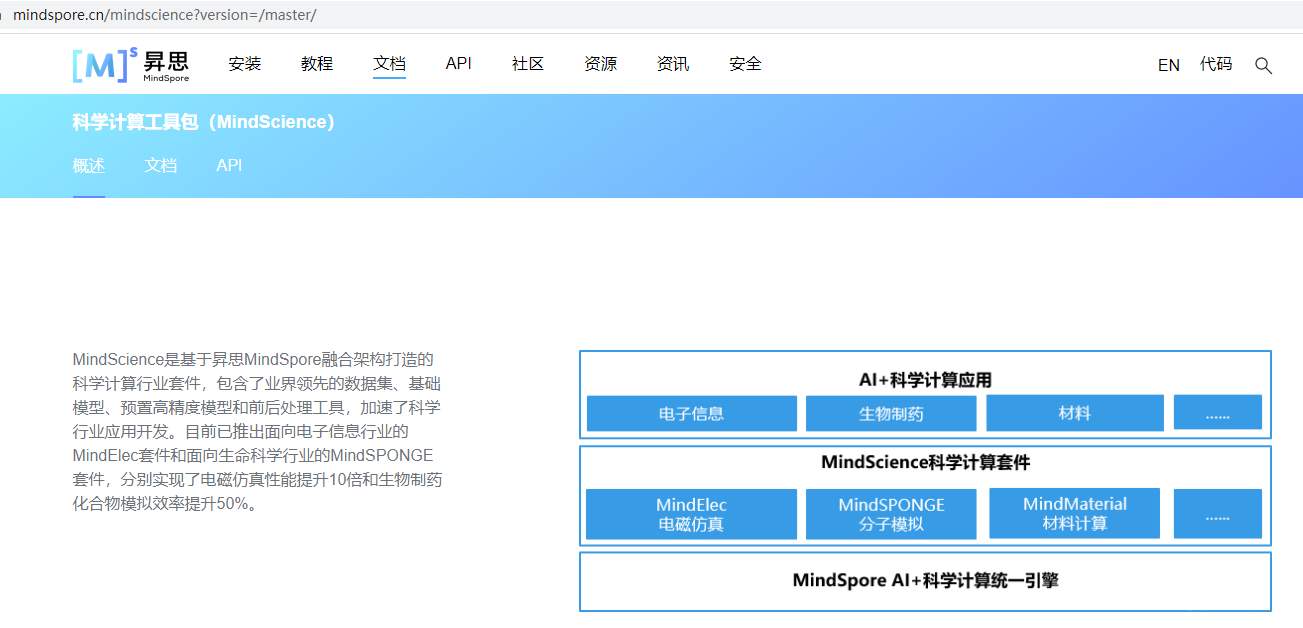
所以,这次专门推出了一个科学计算行业套件,叫做MindScience,( https://mindspore.cn/mindscience?version=/master/ )其实仔细品品,这个Science中文翻译成“昇思”可能才是对的。(张小白你打住好不好?不好意思。。。嘴欠。。。)
当然,MindScience只是个全集,里面还需要根据行业来YoLo一下,目前典型的分两类:MindElec——电磁仿真,MindSPONGE——分子模拟。
其实分子模拟在MindSpore 1.2版本好像就出现过吧?https://zhuanlan.zhihu.com/p/375042410
随着华为跟高校的深度合作,想必这样的分类也会越来越多。
张小白翻了翻官网具体的案例,分子模拟的案例好像都是基于GPU,电磁仿真的案例好像都是基于Ascend。既然张小白暂时用的是wsl的GPU环境,那么就只能先试试MindScience之MindSPONGE了。先阅读后干活,这样可以减少无谓的消耗。。。
https://mindspore.cn/mindscience/docs/zh-CN/master/mindsponge/intro_and_install.html
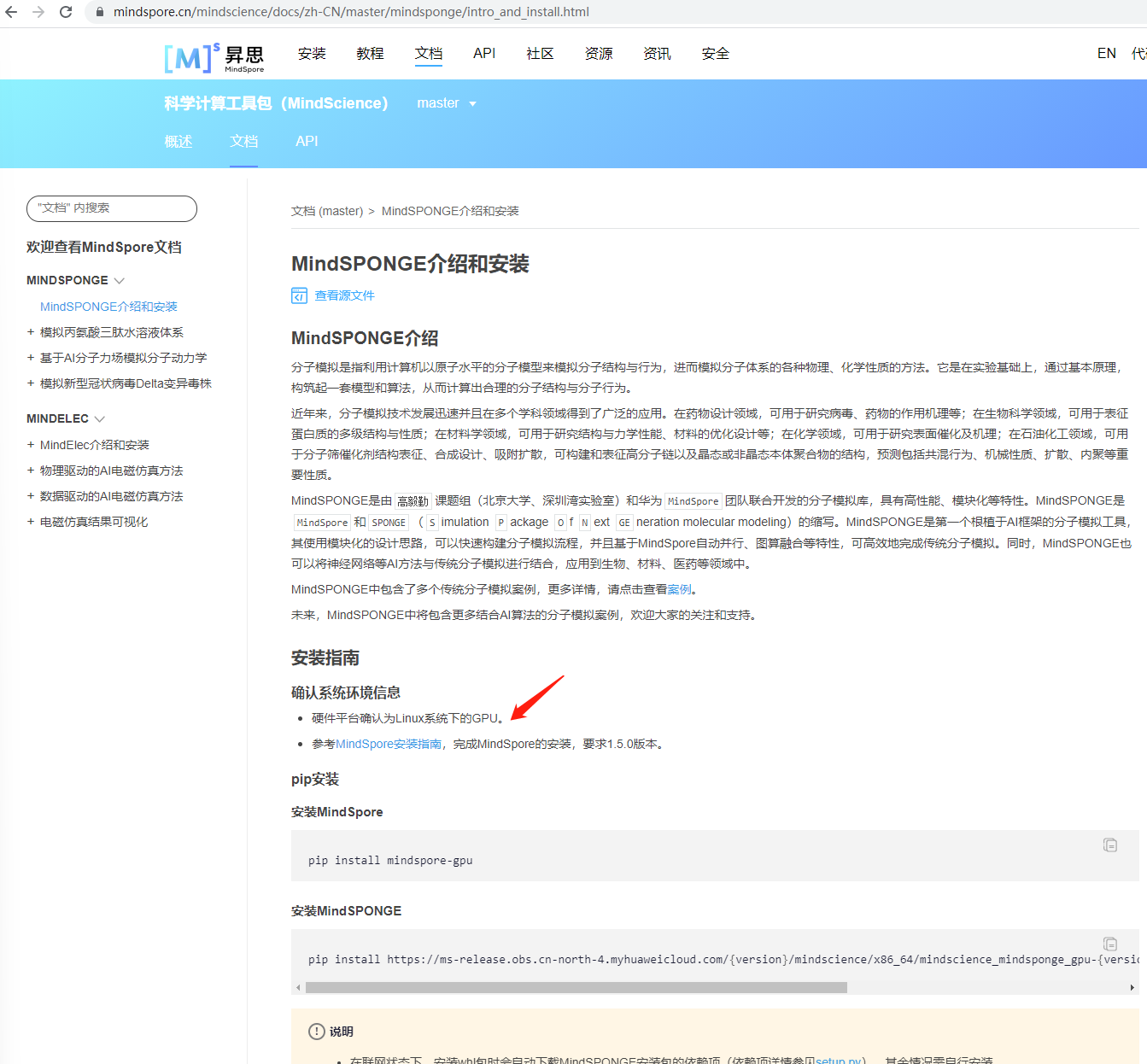
在本文的一开始,mindspore 1.5.0 GPU版本已经装好了。所以下面装MindSPONGE。
根据 https://www.mindspore.cn/news/newschildren?id=734 文档,
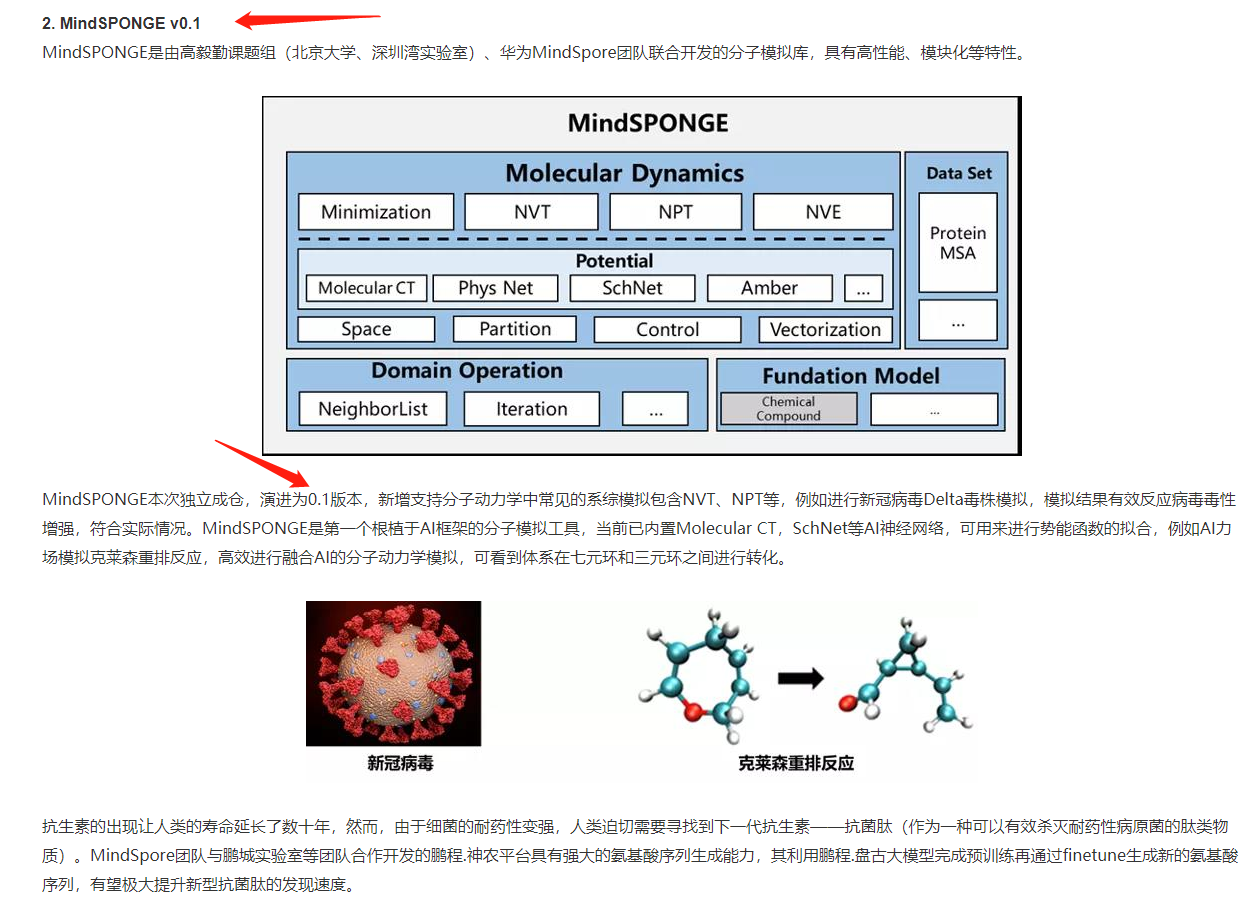
应该下载0.1版本:
打开:https://mindspore.cn/versions 搜索 MindSPONGE:
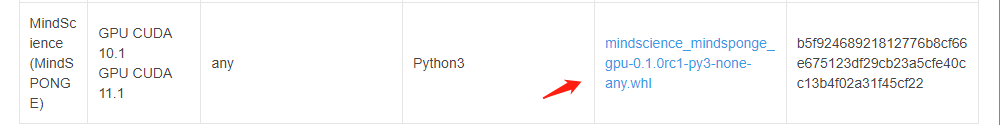
复制上图的链接进行安装:
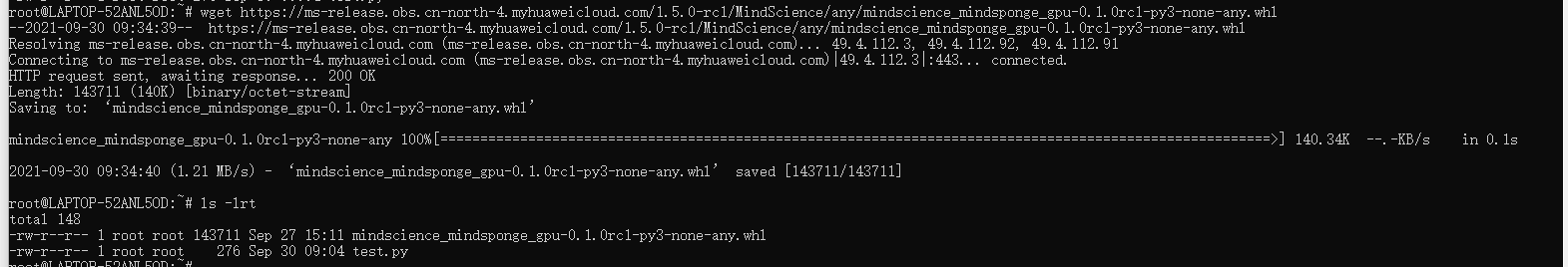
pip install ./mindscience_mindsponge_gpu-0.1.0rc1-py3-none-any.whl -i https://pypi.tuna.tsinghua.edu.cn/simple

验证mindscience安装:
python -c 'import mindsponge'
暂时没报错。
然后开始下载AmberTools。事实证明,这次试验最大的工作量就在这里。
打开:http://ambermd.org/GetAmber.php
打开 http://ambermd.org/InstUbuntu.php
点开 Installation->Ubuntu:
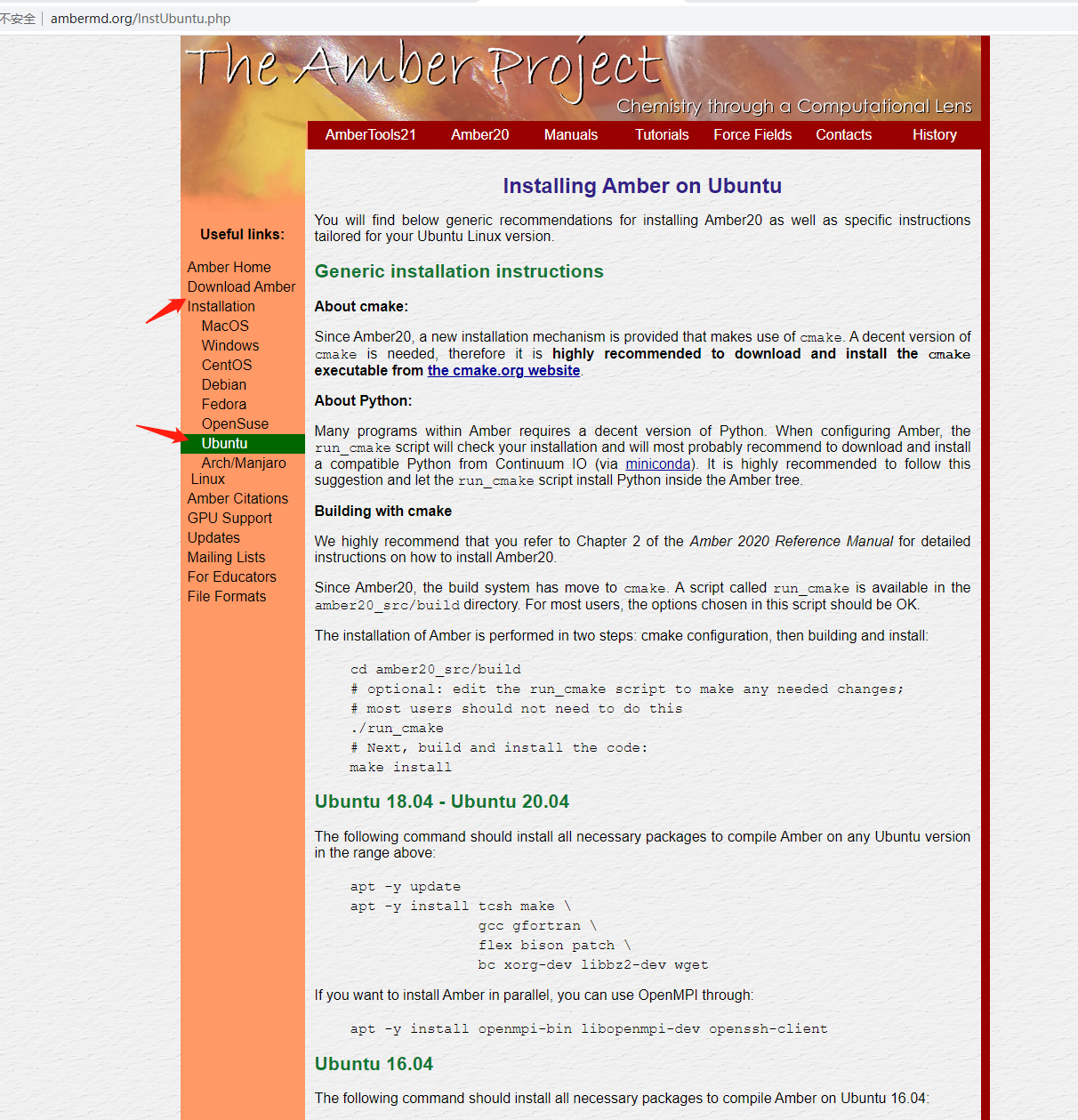
这里告诉了你具体的编译方法。
那么,我们开干吧。
先下载 AmberTools21的源码:
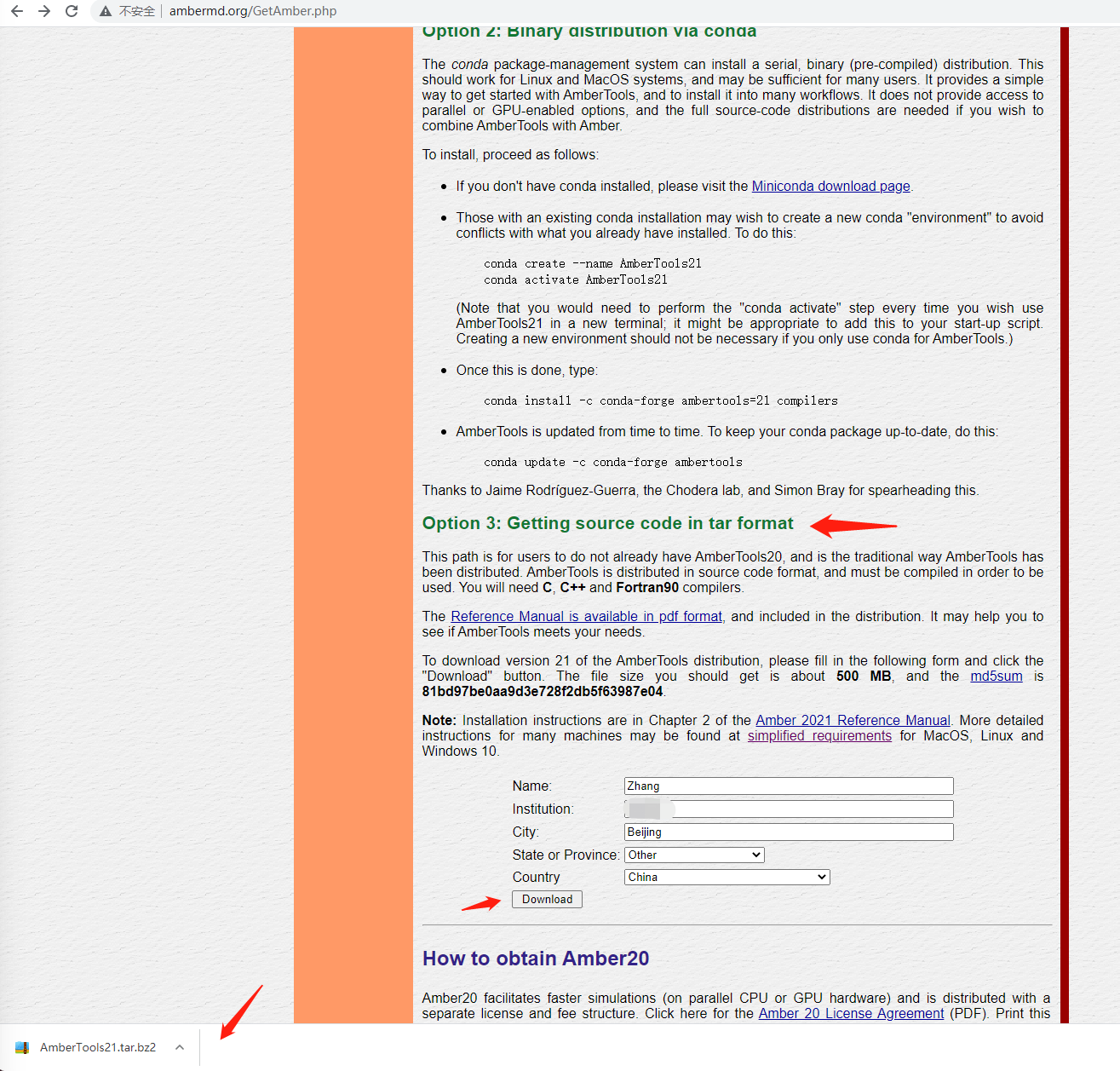
解压:
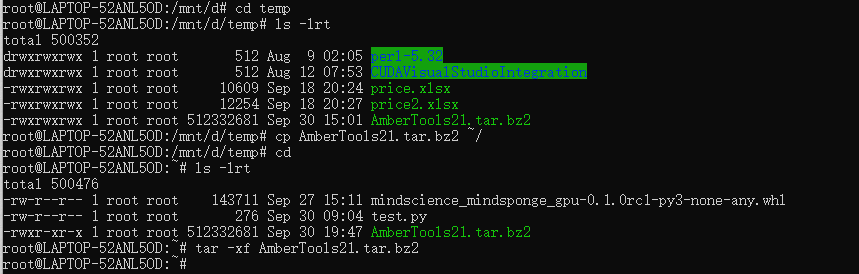
看看目录:
先试着configure GPU版本:
./configure --with-python /root/anaconda3/bin/python -cuda gnu
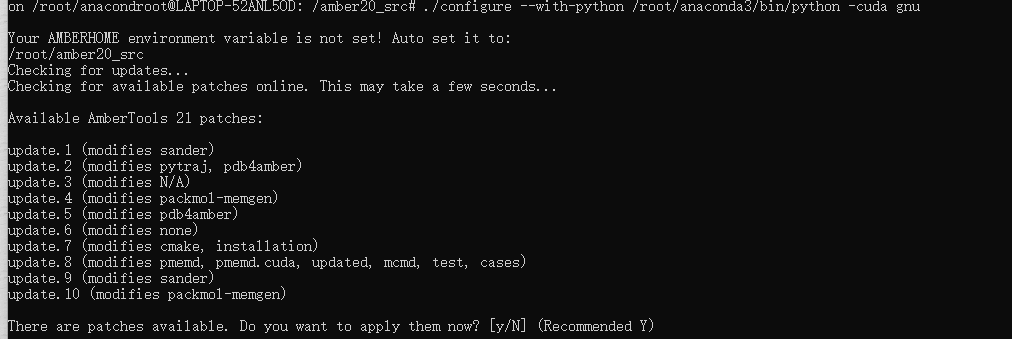
按Y继续:
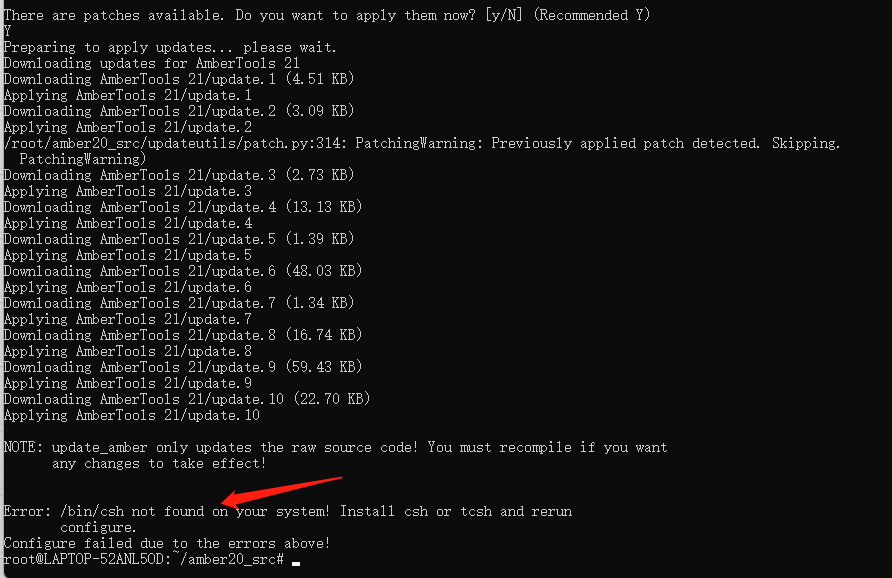
好像报错了,说csh没有。
那就装csh吧:
apt-get install csh
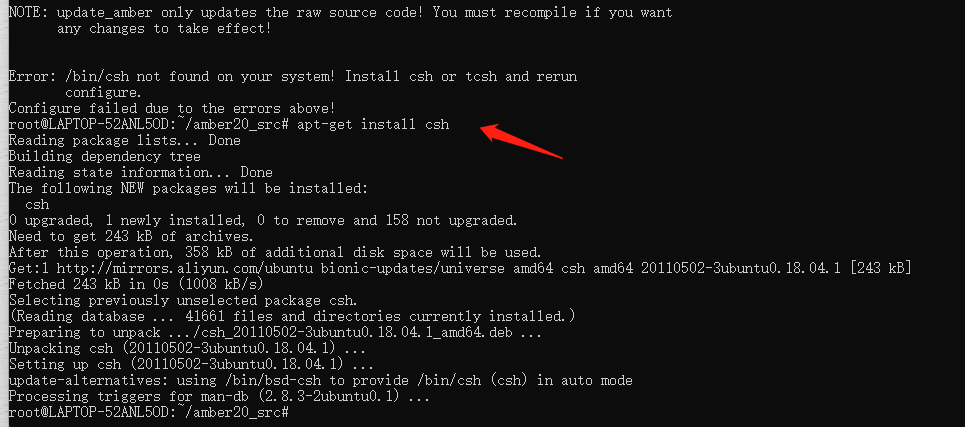
重新configure
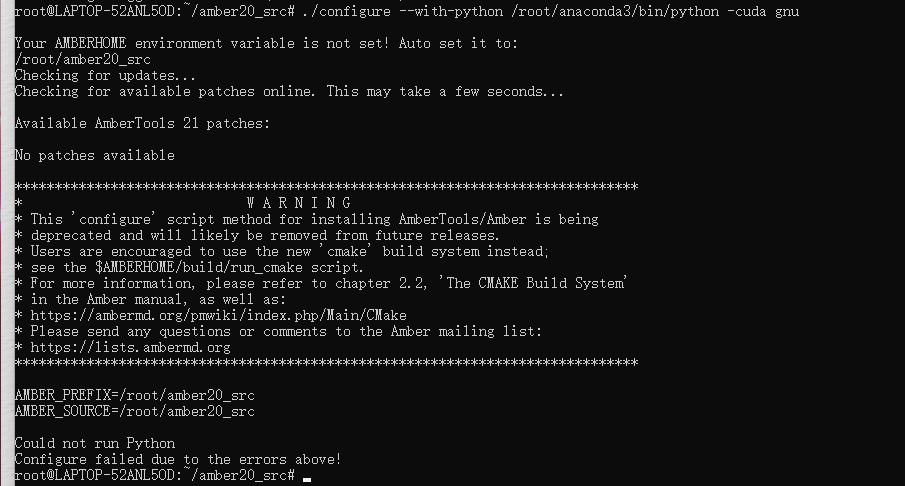
又报错,说Python路径不对,修改为which python的结果:
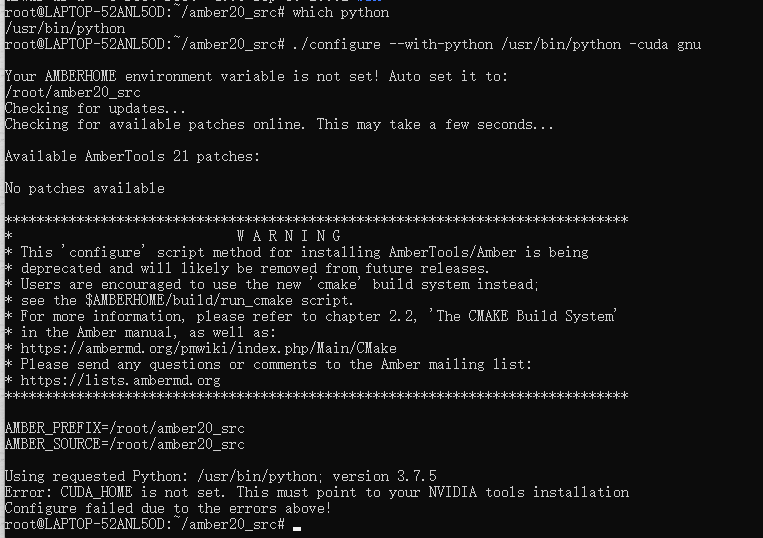
CUDA_HOME没设置,那就设置一下:
export CUDA_HOME=/usr/local/cuda
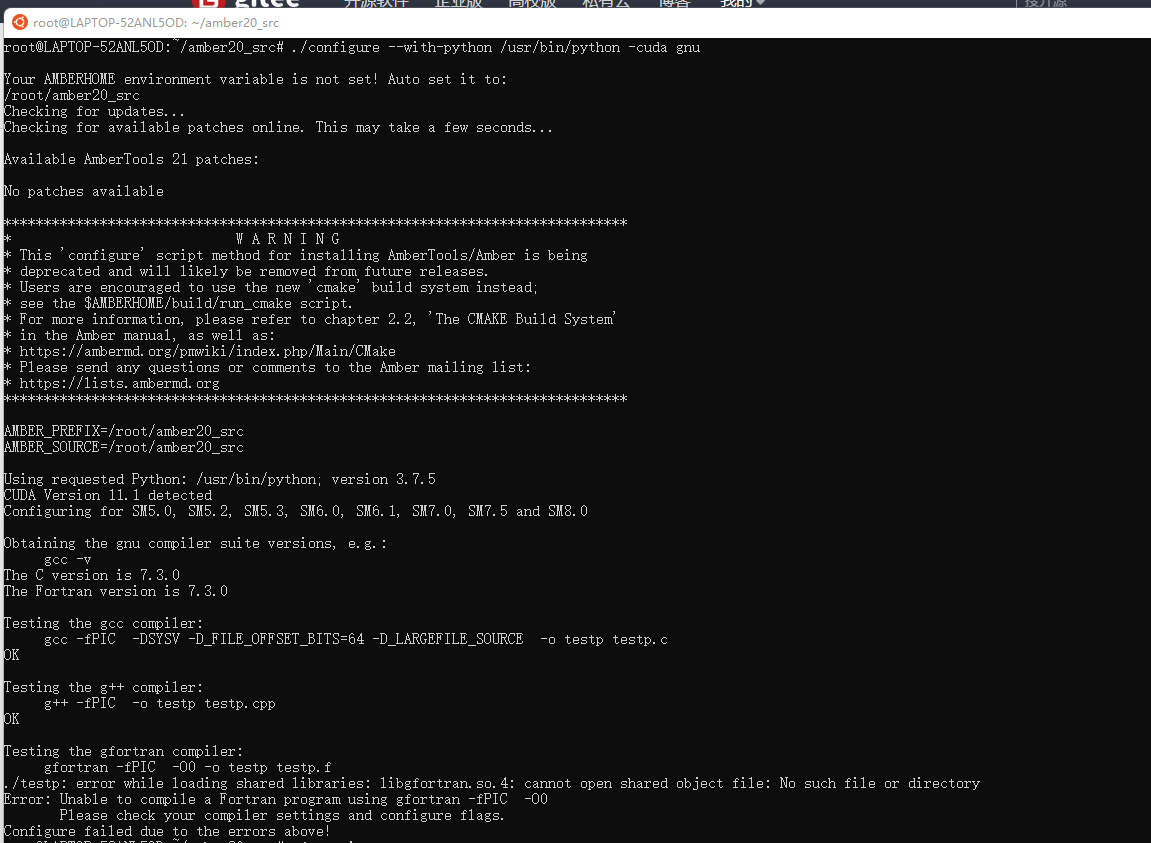
找不到 fortran的so文件。fortran张小白隐约记得是科学计算的语言。。听说比1972年诞生的C语言还要老。查了一下,果然如此,它是1954年提出,1956年开始正式使用。
不过,看来这条路行不通啊。
张小白又开始了度你千遍也不厌倦的过程:
参考 https://www.cnblogs.com/wq242424/p/14283900.html
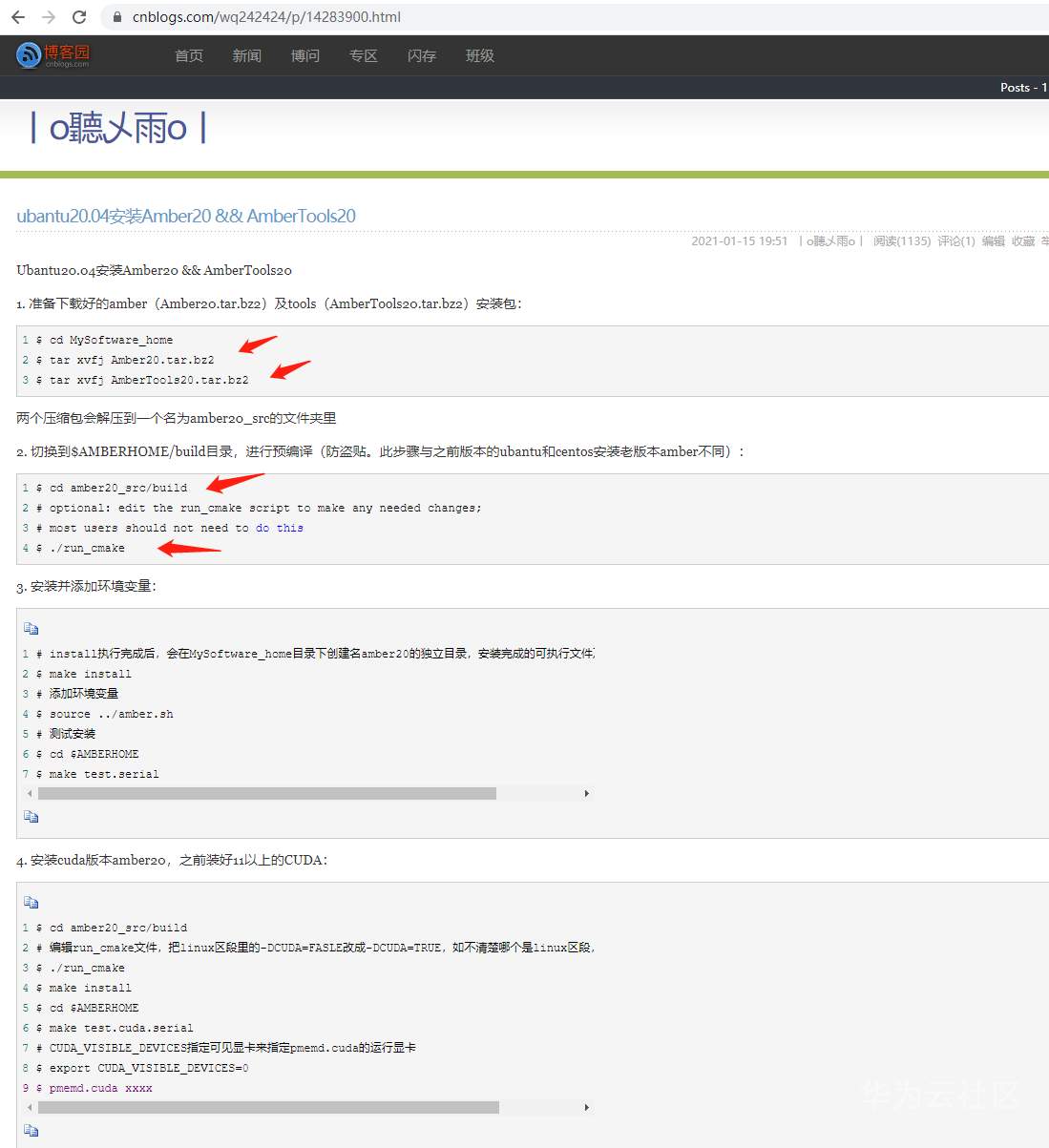
恩,应该是前面编译的姿势不对。
继续开干:
./run_make
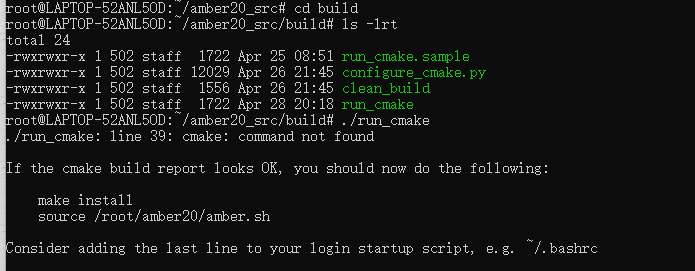
找不到cmake。。。那就装cmake:
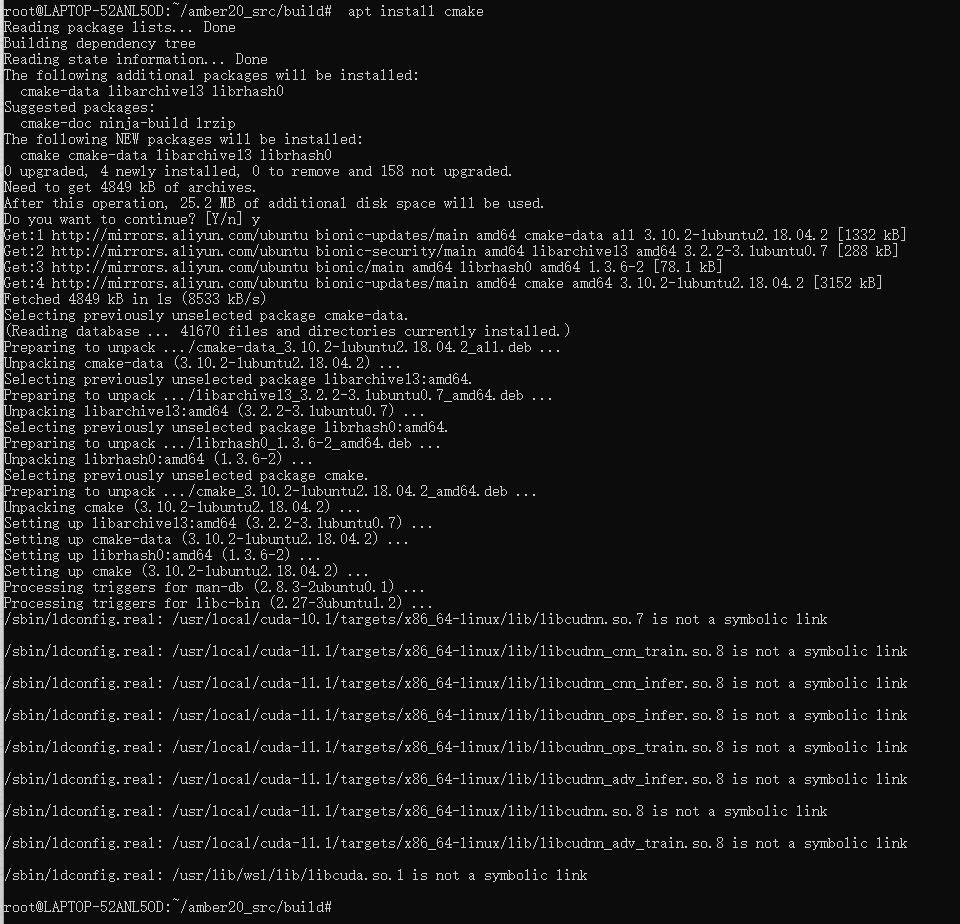
重来:
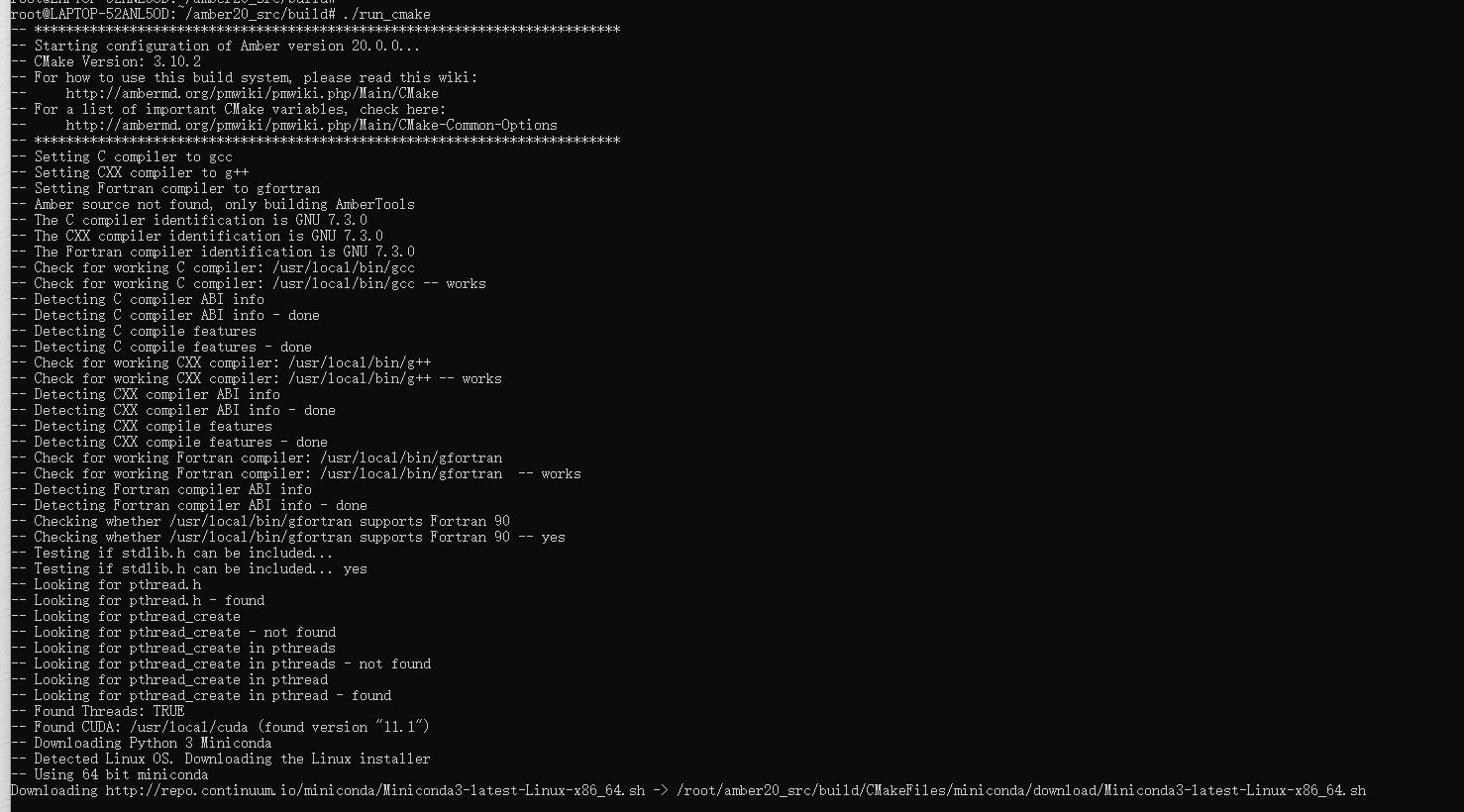
。。。
。。。
...
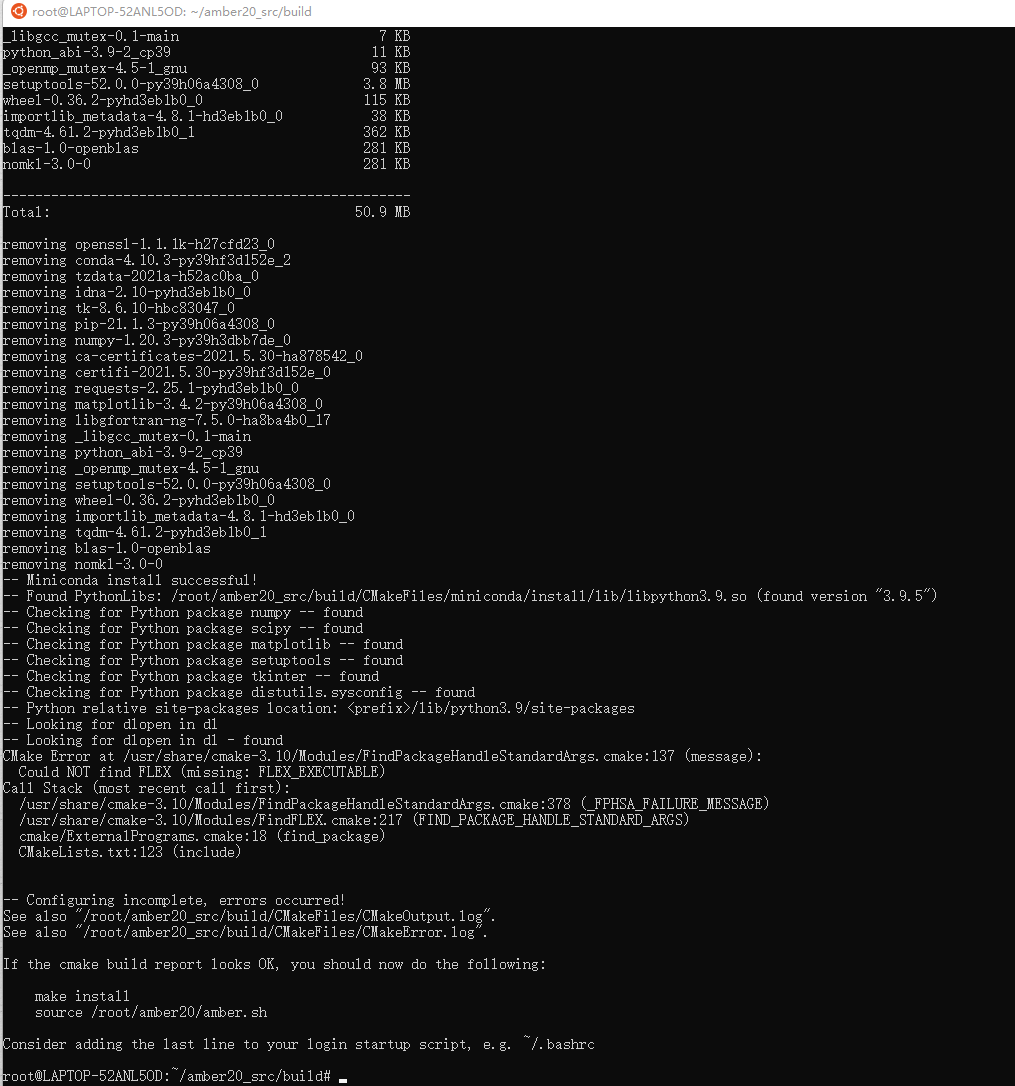
找不到FLEX,参考 https://blog.csdn.net/pas_zoujp/article/details/117351781 ,那就装FLEX,3080你想要啥张小白都给你。。。
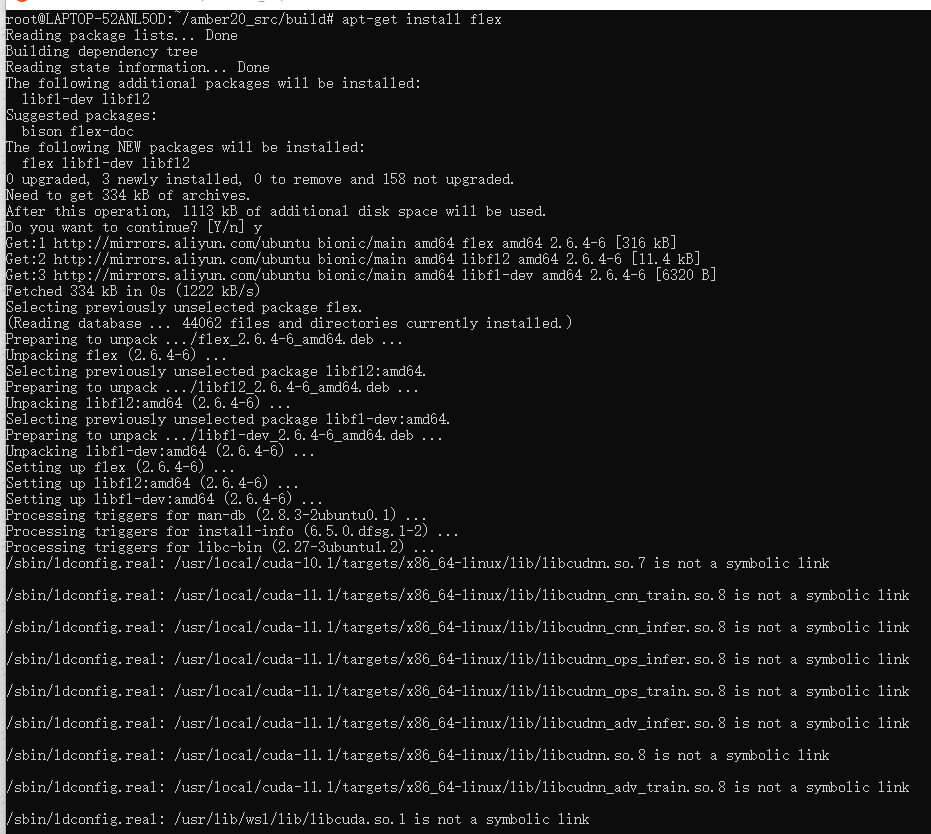
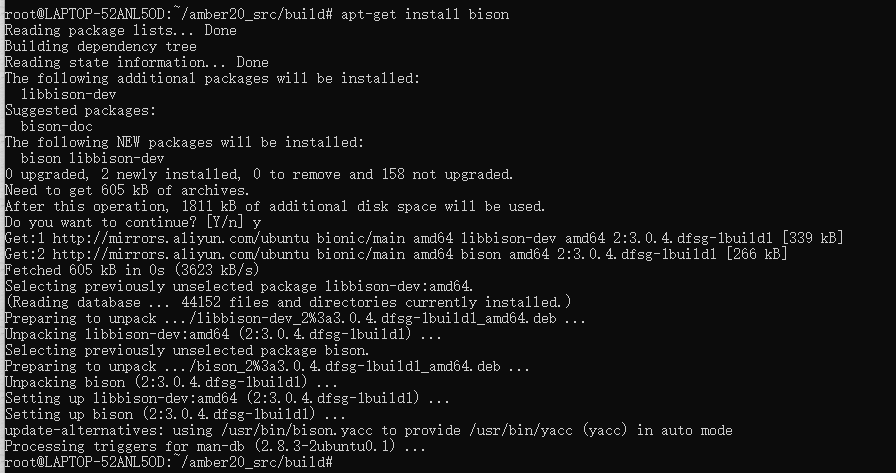
再来:
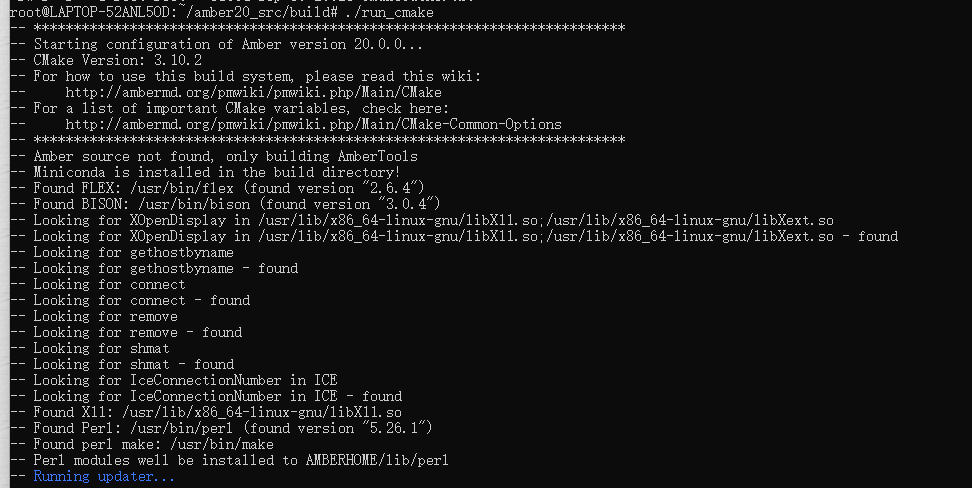
。。
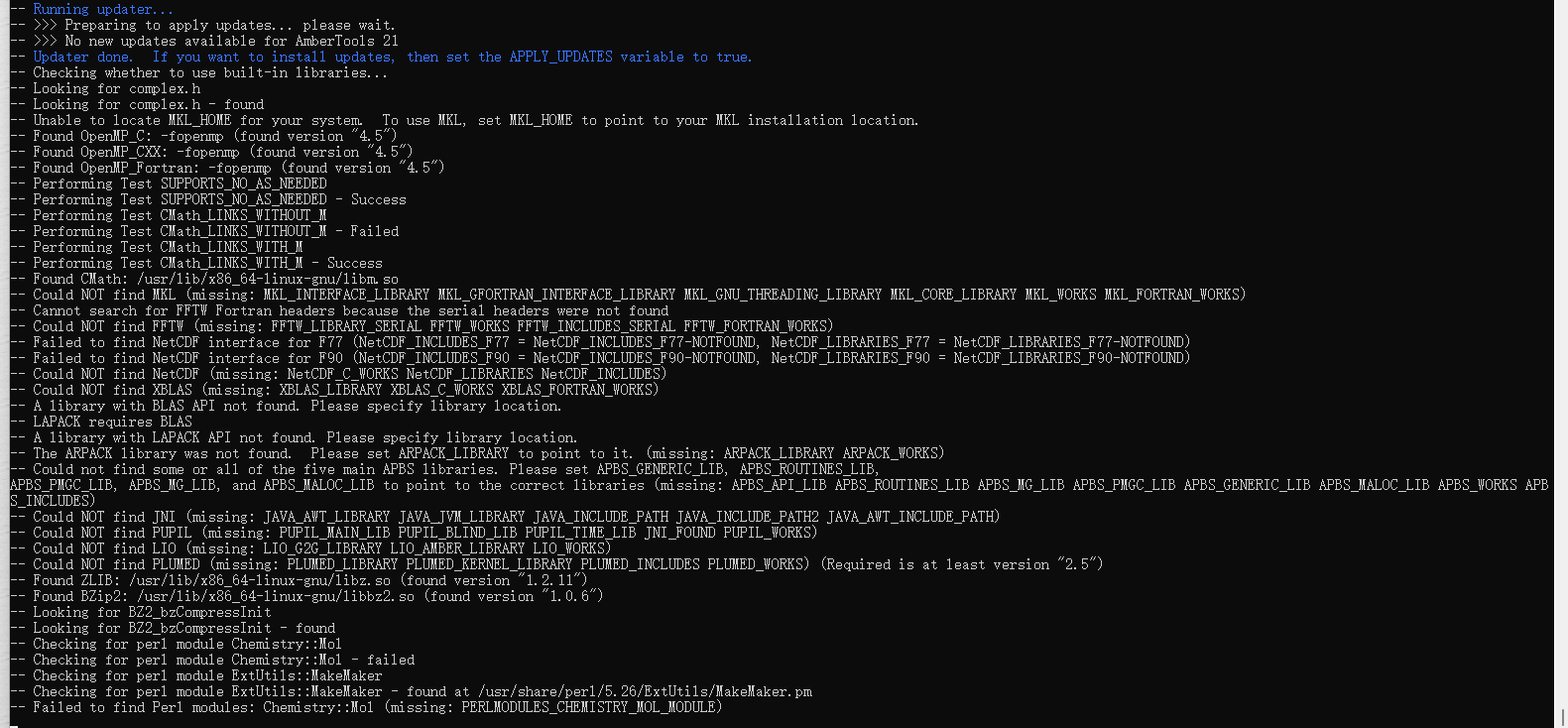
。。。
很好!按照提示继续做下去:
make install
source /root/amber20/amber.sh
...
...
。。。
。。。
恩,成功安装了Amber.
那就开始执行这个tleap工具吧:
tleap:
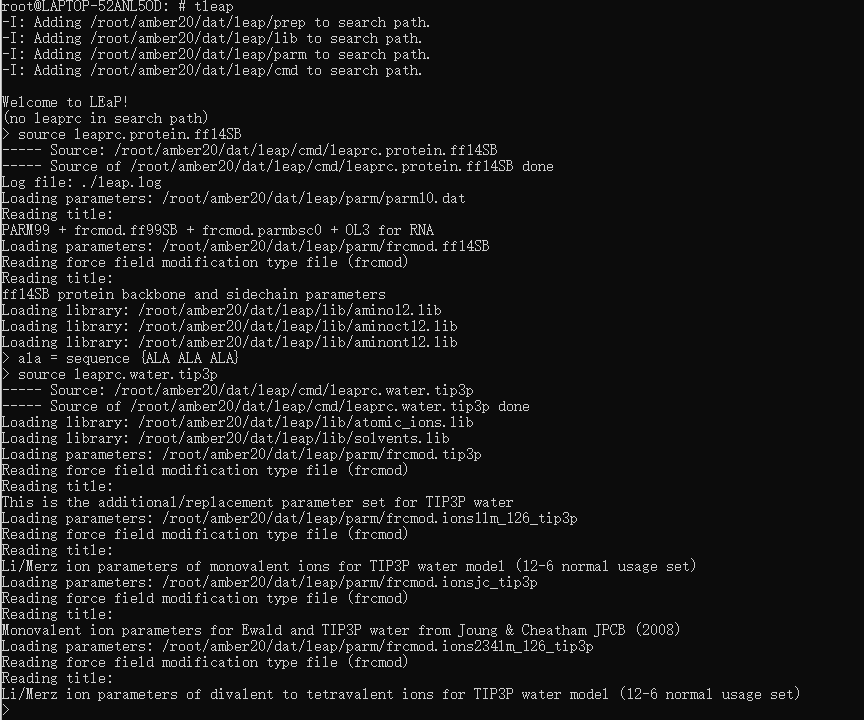
加载tleap自带的ff14SB力场
source leaprc.protein.ff14SB搭建丙氨酸三肽模型
ala = sequence {ALA ALA ALA}利用tleap加载其自带的tip3p力场
source leaprc.water.tip3p
利用tleap中的slovatebox溶解丙氨酸三肽链, 完成体系构建。10.0代表加入的水距离我们溶解的分子及体系边界至少在10.0埃以上
solvatebox ala TIP3PBOX 10.0
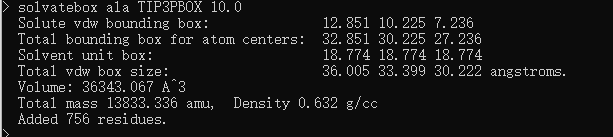
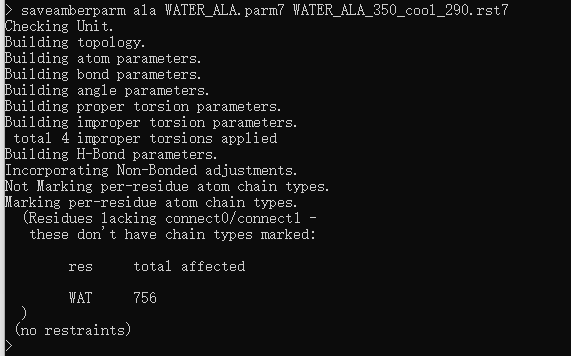
将建好的体系保存成parm7及rst7文件
saveamberparm ala WATER_ALA.parm7 WATER_ALA_350_cool_290.rst7
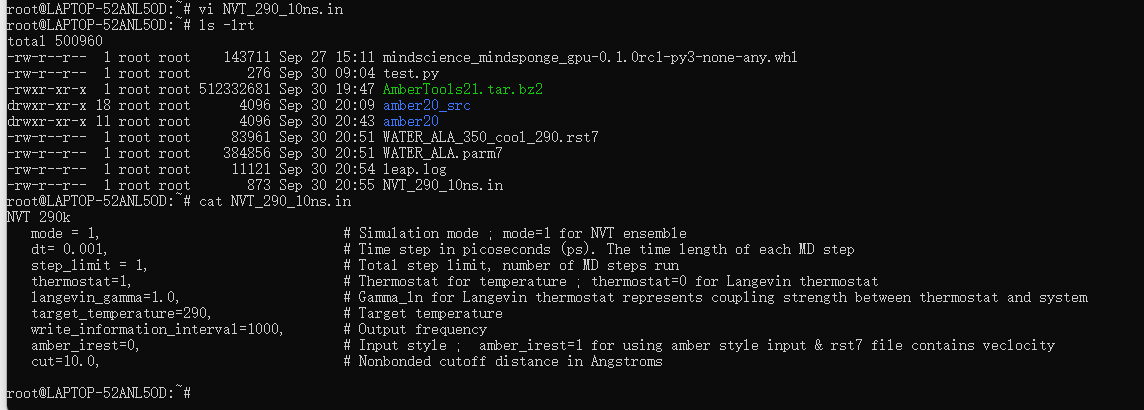
手工编辑 in文件:
加载数据:
myload.py
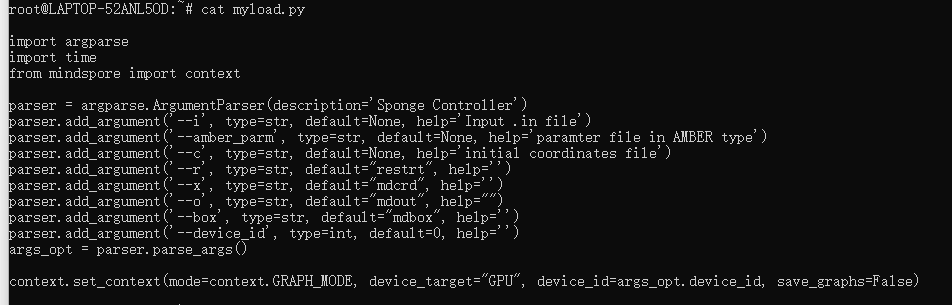
python myload.py
构建模拟流程
mycreate.py
好像不知道怎么玩下去了。
那就到gitee仓库,下载mindscience的码云仓库:
git clone https://gitee.com/mindspore/mindscience.git
切换到 /mindscience/MindSPONGE/examples/polypeptide/scripts 目录
执行 ./run.sh
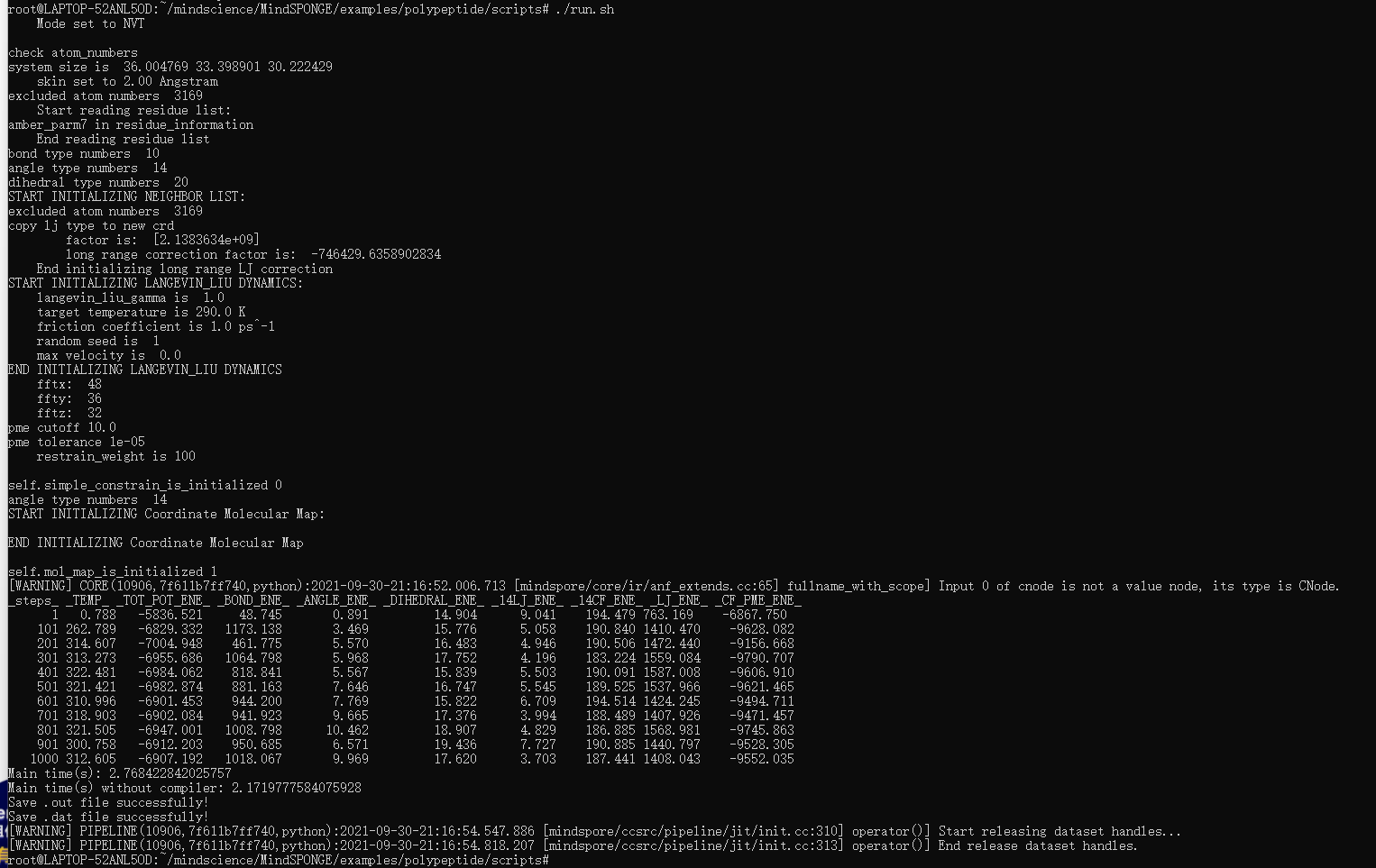
其实run.sh的命令内容如下:
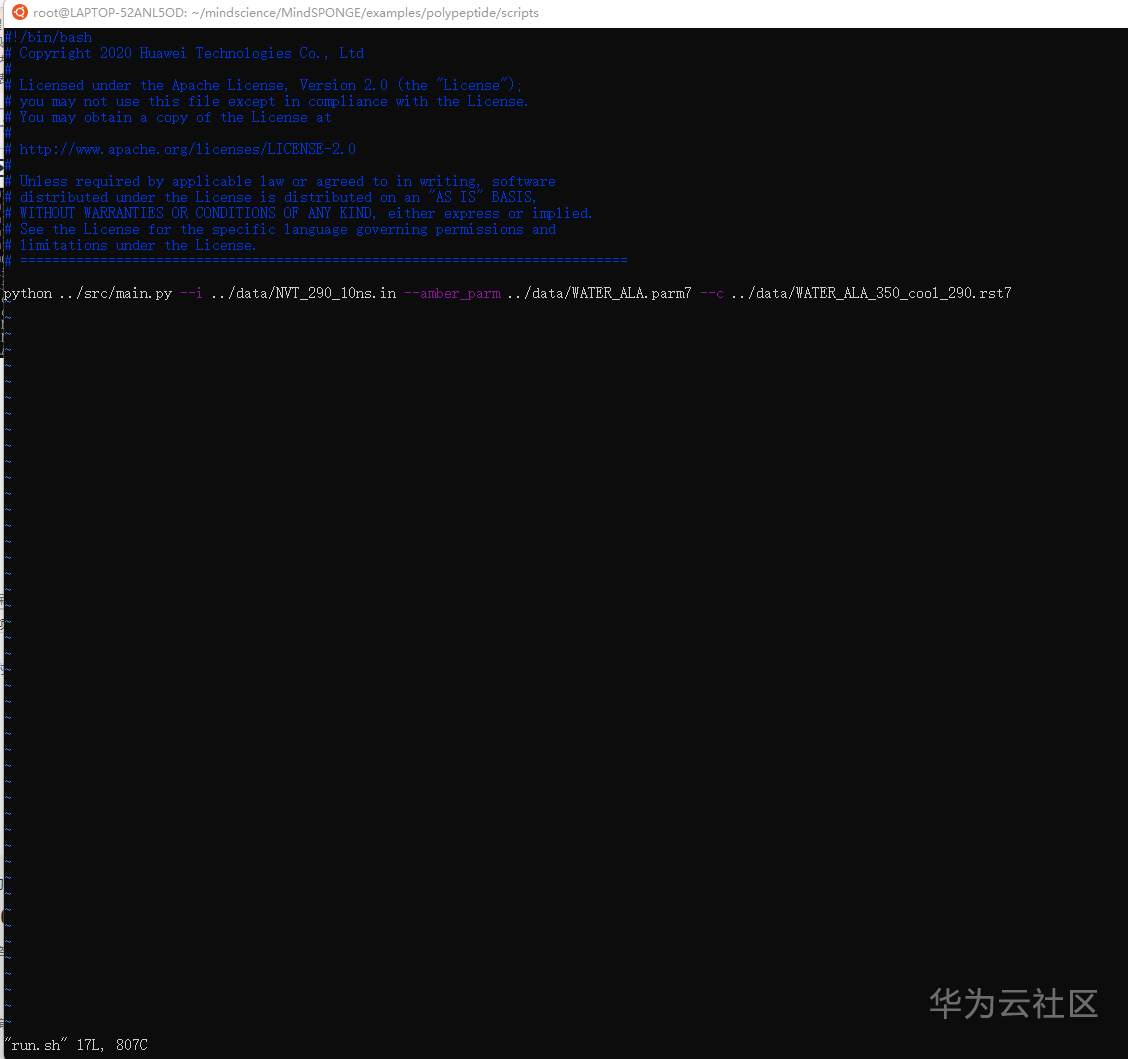
只是它都是执行的是data下的内容。
那我们改写下,用刚才由tleap自己生成的参数:
python ../src/main.py --i /root/NVT_290_10ns.in --amber_parm /root/WATER_ALA.parm7 --c /root/WATER_ALA_350_cool_290.rst7
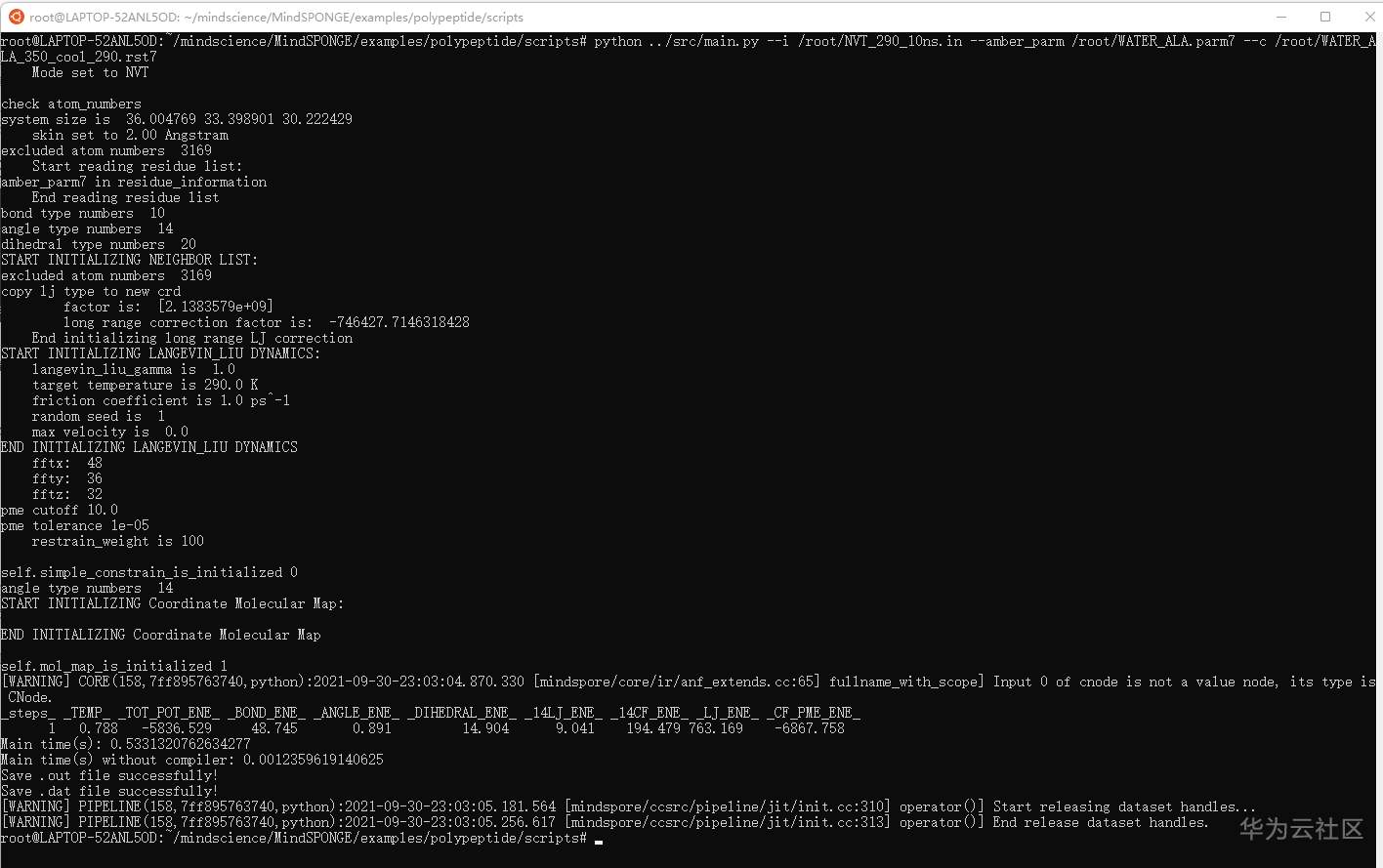
好像输出的结果比 run.sh少几行。
结果贴出来看看:
root@LAPTOP-52ANL5OD:~/mindscience/MindSPONGE/examples/polypeptide/scripts# python ../src/main.py --i /root/NVT_290_10ns.in --amber_parm /root/WATER_ALA.parm7 --c /root/WATER_ALA_350_cool_290.rst7
Mode set to NVT
check atom_numbers
system size is 36.004769 33.398901 30.222429
skin set to 2.00 Angstram
excluded atom numbers 3169
Start reading residue list:
amber_parm7 in residue_information
End reading residue list
bond type numbers 10
angle type numbers 14
dihedral type numbers 20
START INITIALIZING NEIGHBOR LIST:
excluded atom numbers 3169
copy lj type to new crd
factor is: [2.1383579e+09]
long range correction factor is: -746427.7146318428
End initializing long range LJ correction
START INITIALIZING LANGEVIN_LIU DYNAMICS:
langevin_liu_gamma is 1.0
target temperature is 290.0 K
friction coefficient is 1.0 ps^-1
random seed is 1
max velocity is 0.0
END INITIALIZING LANGEVIN_LIU DYNAMICS
fftx: 48
ffty: 36
fftz: 32
pme cutoff 10.0
pme tolerance 1e-05
restrain_weight is 100
self.simple_constrain_is_initialized 0
angle type numbers 14
START INITIALIZING Coordinate Molecular Map:
END INITIALIZING Coordinate Molecular Map
self.mol_map_is_initialized 1
[WARNING] CORE(158,7ff895763740,python):2021-09-30-23:03:04.870.330 [mindspore/core/ir/anf_extends.cc:65] fullname_with_scope] Input 0 of cnode is not a value node, its type is CNode.
_steps_ _TEMP_ _TOT_POT_ENE_ _BOND_ENE_ _ANGLE_ENE_ _DIHEDRAL_ENE_ _14LJ_ENE_ _14CF_ENE_ _LJ_ENE_ _CF_PME_ENE_
1 0.788 -5836.529 48.745 0.891 14.904 9.041 194.479 763.169 -6867.758
Main time(s): 0.5331320762634277
Main time(s) without compiler: 0.0012359619140625
Save .out file successfully!
Save .dat file successfully!
[WARNING] PIPELINE(158,7ff895763740,python):2021-09-30-23:03:05.181.564 [mindspore/ccsrc/pipeline/jit/init.cc:310] operator()] Start releasing dataset handles...
[WARNING] PIPELINE(158,7ff895763740,python):2021-09-30-23:03:05.256.617 [mindspore/ccsrc/pipeline/jit/init.cc:313] operator()] End release dataset handles.其中记录了模拟过程中输出的各类能量, 分别是迭代次数(steps),温度(TEMP),总能量(TOT_POT_E),键长(BOND_ENE),键角(ANGLE_ENE),二面角相互作用(DIHEDRAL_ENE),非键相互作用,其包含静电力及Leonard-Jones相互作用。(这段文字全部是照抄的,因为真的看不懂)
反正这就是张小白使用MindSpore 1.5的MindScience和Amber模拟丙氨酸三肽水溶液体系的过程。至于这个溶液是干嘛的,张小白真的一点兴趣都没有,估计不能喝。还是蜜雪冰城适合张小白。
(全文完,谢谢阅读)
- 点赞
- 收藏
- 关注作者


评论(0)